БИОХИМИЯ И МОЛЕКУЛЯРНАЯ БИОЛОГИЯ - В. ЭЛЛИОТ - 2002
ГЛАВА 20. СИНТЕЗ И РЕПАРАЦИЯ ДНК
Синтез ДНК - сложный процесс, механизмы которого лучше изучены на бактериальных клетках Е. coli, чем на эукариотических. Впрочем, в обоих случаях процессы в основном сходны.
Каждый раз во время деления клетки содержание ДНК должно удвоиться или, как обычно говорят, хромосома(ы) должна(ы) реплицироваться. Тогда каждая дочерняя клетка получает полный набор ДНК. Нет необходимости подчеркивать важность точной репликации, поскольку наличие в гене одного неправильного основания может привести к синтезу белка с нарушенной функцией.
Общие принципы репликации ДНК
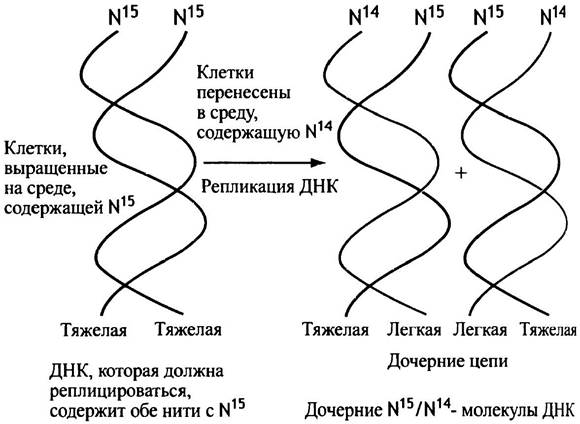
Основу хромосомы составляет одна непрерывная двухцепочечная молекула ДНК. Ее репликация происходит полуконсервативным способом. Две исходные цепи, называемые родительскими, отделяются друг от друга: каждая служит матрицей для синтеза новой цепи, каждая новая двойная спираль содержит одну старую и одну новую цепь. Это было доказано в классическом эксперименте (рис. 20.1).
Рис. 20.1. Доказательство полуконсервативного способа репликации ДНК по Мезельсону и Сталю. ДНК клеток метили путем выращивания на среде, содержащей в качестве источника азота тяжелый изотоп N15; он включался в состав ДНК, и обе родительских нити ДНК получались «тяжелыми». Затем клетки переносили на среду, содержащую обычный природный изотоп N14. Анализ в градиенте плотности показал, что в ходе первого деления после пересева каждая молекула ДНК содержала одну «тяжелую» и одну «легкую» цепь. Появление ДНК с «легкими» и «тяжелыми» цепями указывало на полуконсервативный способ репликации. Продолжение эксперимента в последующих делениях подтвердило этот результат. Цветом выделены вновь синтезированные цепи
В основе образования новой цепи лежит принцип комплементарности оснований (G с С и А с Т), так что последовательность оснований в родительской цепи однозначно определяет последовательность оснований в новой - дочерней - цепи.
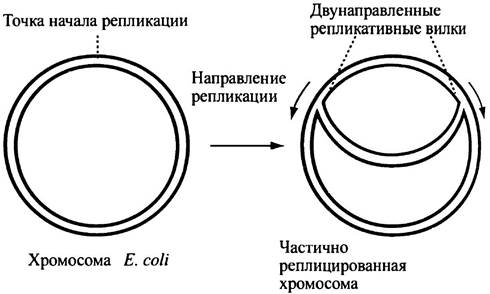
Поскольку в ДНК пары оснований Уотсона-Крика образуют водородные связи, синтез новых молекул может произойти только при расхождении цепей, что делает основания доступными для новых взаимодействий с комплементарными основаниями. Хромосома E. coli имеет кольцевую форму и содержит около четырех миллионов пар оснований. В определенной точке, называемой точкой начала репликации, цепи расходятся и образуются две репликативные вилки, которые движутся в противоположных направлениях. ДНК синтезируется со скоростью примерно 500 пар оснований в секунду, причем разделение родительских ДНК и синтез новых цепей происходит одновременно (рис. 20.2). Две вилки встречаются на противоположной стороне кольца. В результате репликации два новых кольца оказываются сцепленными друг с другом. Этот факт представляли доводом против описанной выше модели репликации ДНК. Остается лишь удивляться проницательности Крика, утверждавшего, что эволюция позаботилась о механизме, разделяющем кольца. Впоследствии выяснилось, что эту функцию выполняет фермент топоизомераза II (см. с. 245).
Рис. 20.2. Двунаправленная репликация хромосомы Е. coli. Вновь синтезированные нити выделены цветом
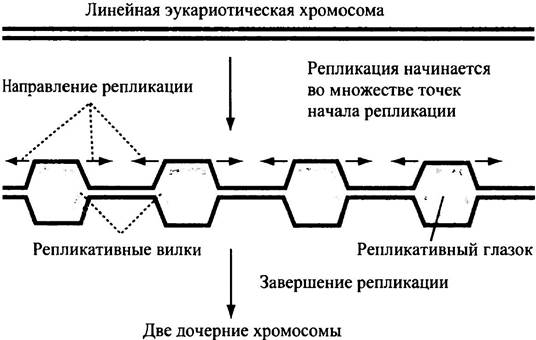
У эукариот синтез ДНК в репликативной вилке идет со скоростью примерно 50 пар оснований в секунду - слишком медленно для того, чтобы в хромосоме за время, отведенное на клеточное деление, синтезировалась ДНК огромных размеров. На самом деле репликация эукариотической ДНК начинается одновременно во многих точках (их, вероятно, более тысячи). От каждой такой точки в обоих направлениях движутся две репликативные вилки (рис. 20.3); в остальном процесс протекает аналогично репликации у Е. coli.
Рис. 20.3. Схематичное изображение, демонстрирующее образование множества двунаправленных репликативных вилок в эукариотической хромосоме
Регуляция инициации репликации ДНК в Е. coli
Перед делением клетки Е. coli должно произойти полное удвоение хромосомы; само клеточное деление протекает позже, минут через 20. Точно не известно, каким образом скоординировано клеточное деление и репликация ДНК. Для осуществления этих процессов необходимы синтез белка и значительное увеличение размера клетки. Ранее было показано (см. рис. 20.2), что у E. coli существует единственная точка начала репликации ДНК (oriC), откуда в двух направлениях идет репликация. Целую хромосому E. coli называют репликоном; у эукариот репликон - отрезок ДНК, репликация которого протекает под контролем единственной точки начала репликации.
Точка начала репликации имеет специфическую последовательность оснований, богатую парами А-Т, что, вероятно, облегчает разделение нитей (напомним, пары А-Т имеют две водородные связи, а пары G-С три, поэтому первые слабее связаны друг с другом). Во время инициации к этой области присоединяется множество молекул белка, названного DnaA, после чего происходит отделение нитей. Это, в свою очередь, помогает основному расплетающему цепи ферменту хеликазе, работающему в каждой репликативной вилке, начинать и продолжать последовательное расплетание цепей в обоих направлениях. О хеликазе (называемой также DnaB, т. е. белком, кодируемым геном dnaB) будет рассказано при описании механизма синтеза ДНК.
Инициация репликации ДНК у эукариот
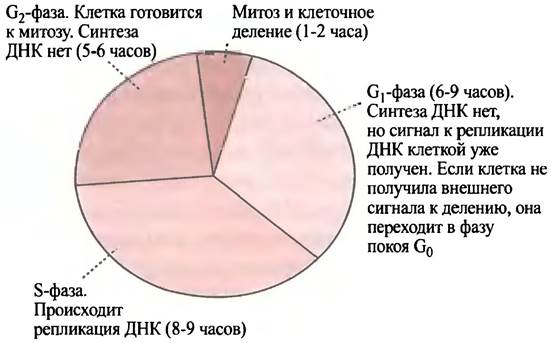
Эукариотические клетки имеют более сложный клеточный цикл, чем клетки Е. coli, и в них синтез ДНК приурочен к определенному периоду, который называется S-фазой (от англ. synthesis phase - фаза синтеза) (рис. 20.4). Продолжительность клеточных циклов различных клеток сильно различается; в культуре клеток животных из общего 24-часового клеточного цикла S-фаза занимает около 8 часов. S-фазе предшествует G1-фаза (от англ. gap in DNA synthesis - промежуток в синтезе ДНК). Чтобы приступить к делению, клетки млекопитающих должны получить извне соответствующий сигнал. Таким сигналом служат белковые молекулы - факторы роста.
Рис. 20.4. Цикл эукариотической клетки. Продолжительность клеточного цикла разная у различных типов клеток. Здесь приведены фазы цикла для быстроделящейся культуры клеток млекопитающих (полный цикл 24 часа)
На поверхности клеток они связываются с рецепторами, которые посылают сигнал к делению внутрь клетки (см. главу 26). Как уже отмечалось, в хромосоме животных имеется множество точек начала репликации, от которых репликация идет в двух направлениях. Пока неизвестно, как регулируется инициация репликации. Существенно, что каждый репликон в ходе одного клеточного деления активируется только один раз.
Расплетание двойной спирали ДНК и сверхспирализация
Разделение цепей ДНК с помощью хеликазы представляет собой топологическую (физическую) проблему. Для объяснения этого явления мы должны познакомиться с понятием сверхспирализации ДНК.
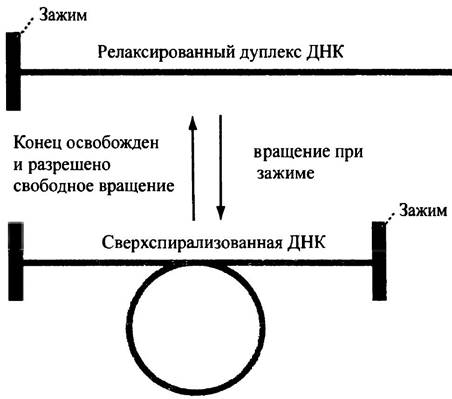
Две цепи ДНК (дуплекс ДНК) образуют правозакрученную спираль, на один виток которой приходится 10 пар оснований. Изолированный короткий фрагмент линейной ДНК, который может легко вращаться вокруг своей длинной оси, спонтанно принимает свободную конфигурацию, известную как релаксированная форма. Представим, что один конец дуплекса не может вращаться, а на противоположном конце образуются дополнительные витки. В этом случае спираль начинает сжиматься, а количество оборотов на этом отрезке ДНК возрастает, в то время как количество пар оснований, приходящихся на один оборот, уменьшается. Это явление называется положительной сверхспирализацией, или сверхскрученностью. Если же поворачивать дуплекс в противоположном направлении, спираль раскручивается: количество оборотов на единицу длины начинает уменьшаться, а количество оснований в одном витке будет возрастать. ДНК перейдет в слабоскрученное состояние, или состояние отрицательной сверхспирализации. Обе формы - и сверхскрученная, и слабо- скрученная - представляют напряженное состояние молекулы, для выхода из которого необходимо закручивание двойной спирали ДНК самой на себя с образованием спирализованной спирали, или сверхспирали (рис. 20.5). Сверхспирализацию легко представить, взяв кусочек дважды закрученного каната. Пусть кто-нибудь держит один конец каната или закрепит его так, чтобы он не мог свободно вращаться, и крутит канат вдоль оси. Образуется спираль, закрученная в соответствии с напряжением вращения. Если отпустить конец сверхспирализованного каната, он немедленно примет исходную (релаксированную) форму.
Рис. 20.5. Схематичное изображение, поясняющее, как закручивание фрагмента ДНК, не имеющего свободы вращения, приводит к сверхспирализации, устраняющей напряжение вращения. Клеточная ДНК прочно фиксирована и не обладает свободой вращения. Впрочем, если свободное вращение станет возможным, сверхспираль перейдет в релаксированное состояние. Если вращение происходит в направлении раскручивания, сверхспираль будет отрицательной; если вращение производить в противоположном направлении, сверхспираль будет положительной (различия между этими двумя формами не приведены)
Что происходит с ДНК во время репликации?
Ведь в клетке ее нить не может свободно вращаться относительно своей оси. У Е. coli замкнутое кольцо хромосомы прочно «фиксирует» ДНК. У эукариот огромная молекула ДНК может образовывать петли (см. рис. 19.7, г), закрепленные белковыми структурами; при этом возникает подобие кольцевых структур, и в результате участки становятся неспособными к свободному вращению. Но при разделении нитей ДНК обязательно происходит вращение дуплекса. Это вызывает сверхспирализацию, которая приводит к появлению положительных сверхвитков в начале репликационной вилки; и так как спираль сжимается, дальнейшее расплетание становится затруднительным. Если напряжение не исчезнет, то оно будет препятствовать расплетанию нитей, и репликация ДНК остановится.
В этом вас убедит очень простой эксперимент. Возьмем короткий кусок двунитевого каната и потянем его концы в разные стороны: канат будет быстро раскручиваться без накопления положительных сверхвитков. При этом нити каната легко и полностью отделятся друг от друга. Теперь свернем длинный кусок такого же каната в кольцо или закрепим один его конец таким образом, чтобы канат не мог свободно вращаться, и попытаемся развести его нити. Положительные супервитки быстро «заклинят» образовавшуюся вилку и воспрепятствуют последующему разделению. То же самое будет происходить при репликации ДНК в клетке, если что-либо не помешает этому
Отсюда следует, что для продолжения синтеза ДНК положительные сверхвитки в начале репликационной вилки должны ослабляться. Это по мере необходимости происходит за счет кратковременных расщеплений полинуклеотидной цепи.
Как устраняются положительные сверхвитки перед репликативной вилкой?
Этот процесс катализирует группа ферментов - топоизомераз. Они действуют на ДНК, изомеризуя или меняя ее топологию. Существует два класса топоизомераз (тип I и II). Сначала мы рассмотрим принцип их действия, а затем объясним их роль в репликации ДНК.
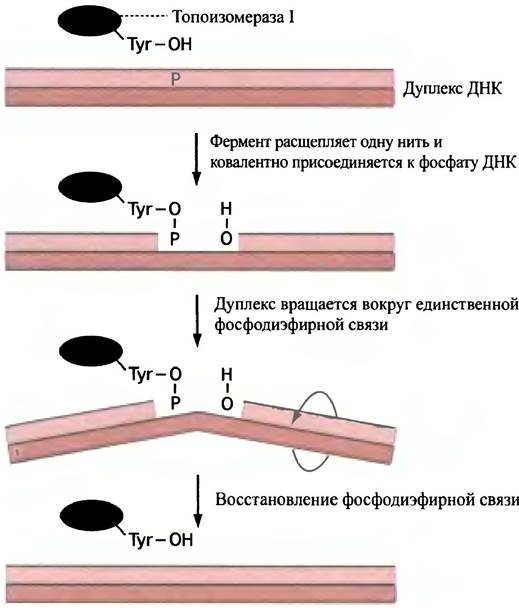
Ферменты типа I расщепляют одну из двух цепей сверхспирализованной двойной спирали, что позволяет всему дуплексу вращаться вокруг единственной фосфодиэфирной связи противоположной цепи, которая в молекуле ДНК играет роль эффективного шарнира. После окончания вращения фермент восстанавливает дуплекс (рис 20.6). Очень важно, что фермент не гидролизует фосфодиэфирную связь, которую атакует. Он просто переносит ее от 3'-ОН группы дезоксирибозы к ОН-группе одного из тирозинов, входящих в состав молекулы фермента. Процесс легко обратим, поскольку при разрезании не происходит потери энергии. Отметим, что топоизомераза I не использует энергию АТР. Сверх- спирализованная молекула ДНК находится в состоянии напряжения, и уровень энергии будет выше, чем в релаксированной форме, вне зависимости от вида сверх- спирализации - положительной или отрицательной.
Рис. 20.6. Реакция, катализируемая топоизомеразой I. Туr-ОН - остаток тирозина в молекуле фермента
Топоизомераза I способна только релаксировать сверхспирализованную ДНК, но не способна ввести новые витки. В то же время в Е. coli имеется особая топоизомераза I, которая может релаксировать только отрицательную сверхспираль ДНК. Впрочем, это не решает проблему раскручивания молекулы, хотя и играет важную роль.
Релаксация положительной сверхспирали, образующейся впереди репликативной вилки Е. coli, происходит путем активного введения отрицательных сверхвитков, которые «нейтрализуют» положительные сверхвитки. Это происходит под действием топоизомеразы II.
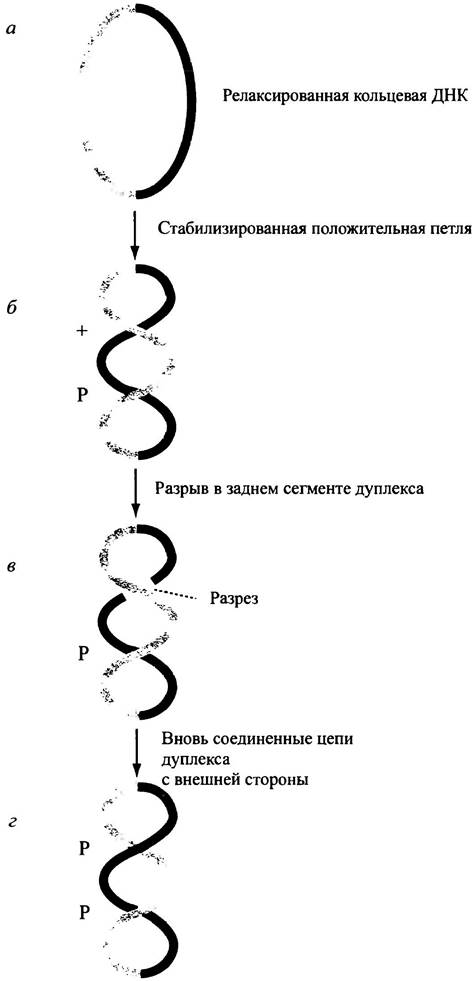
Топоизомераза II разрывает обе нити ДНК в двойном дуплексе, перенося связи на себя, что делает процесс разрыва полинуклеотидных цепей легко обратимым. Кольцевой дуплекс ДНК фермент физически переносит между концами разрыва без вращения цепей. Для этого требуется энергия АТР; возможно, именно благодаря его гидролизу происходят конформационные изменения в белке. У Е. coli топоизомераза II, называемая гиразой, вводит в ДНК отрицательные сверхвитки (рис. 20.7). Для простоты на рисунке изображено образование отрицательных сверхвитков в релаксированной циклической ДНК (см. рис. 20.7, а). Но этот же принцип применим к ДНК в начале репликативной вилки у Е. coli, где возникает положительная сверхспирализация в результате расплетания нитей ДНК. Установлено, что включение отрицательных сверхвитков в этой области соответствует релаксации положительной сверхспирали. Гираза взаимодействует с ДНК таким образом, что последняя наматывается вокруг белка. При этом возникает сверхскручивание (положительная сверхспирализация) в тех местах молекулы, которые связаны с белком (см. рис. 20.7, б). Однако до тех пор, пока не происходит разрывов ковалентных связей, не нарушается и целостность структуры, а, следовательно, локальная положительная сверхспираль, образовавшаяся в ответ на включение белка, может уравновешиваться отрицательной сверхспирализацией где-нибудь в другом месте молекулы ДНК: в результате сверхспирализация сводится к нулю. Затем фермент разрывает обе нити ДНК (см. рис. 20.7, в) и переносит двойную нить с внутренней стороны на внешнюю, после чего скрепляет оба разрыва, превращая положительную петлю в отрицательную (см. рис. 20.7, г). Иными словами, происходит вставка отрицательного сверхвитка. Физическое перемещение ДНК совершается за счет гидролиза АТР. Состояние отрицательной сверхспирали также является напряженным; следовательно, введение отрицательных сверхвитков относится к энергозависимым процессам. Установлено, что расщепление связей в ДНК происходит не путем гидролиза, а в результате временного переноса однонитевых концевых фрагментов ДНК на фермент без потери свободной энергии; поэтому процесс разрыва связей легко обратим.
Рис. 20.7. Реакция включения отрицательных сверхвитков в кольцевую ДНК, катализируемая гиразой. Этот процесс может превращать положительно сверхскрученную ДНК в отрицательно сверхскрученную ДНК
Какова биологическая роль топоизомераз?
С эволюционной точки зрения для выживания клеток чрезвычайно важно решение топологических проблем, и оно происходит с помощью двух типов топоизомераз (I и II), обладающих различными механизмами действия. У многих организмов свойства этих ферментов несколько отличаются. Описано по меньшей мере 11 разновидностей топоизомераз.
У Е. coli топоизомераза I релаксирует только отрицательные сверхспирали. В то же время гираза (топоизомераза II) активно включает отрицательные сверхвитки, и поскольку это приводит к релаксации положительной сверхспирали, тем самым обеспечивается продолжение синтеза ДНК.
У эукариот топоизомераза I может релаксировать оба типа сверхспиралей - и положительную, и отрицательную, а топоизомераза II - только положительную, но не может вводить отрицательные сверхвитки (табл. 20.1). Таким образом, при репликации ДНК у про- и эукариот спутывание цепей при их расплетании предотвращается введением положительных сверхвитков.
Таблица 20.1. Топоизомеразы Е. coli и эукариот: типы и механизм действия
Тип* |
Действие на ДНК |
Эффект на сверхспирализацию |
Топоизомераза I |
||
Е. соli |
Разрывает одну нить ДНК |
Релаксирует отрицательную сверхспирализацию |
Эукариоты |
Разрывает одну нить ДНК |
Релаксирует положительную и отрицательную сверхспирализацию |
Топоизомераза II(гираза) |
||
Е. соli |
Разрывает обе нити ДНК; АТР-зависимая |
Релаксирует положительные сверхвитки; включает отрицательные сверхвитки; разделяет сцепленные кольцевые молекулы ДНК |
Эукариоты |
Разрывает обе нити ДНК; АТР-зависимая |
Релаксирует положительные сверхвитки, но не может вводить отрицательные сверхвитки |
* Топоизомеразы обоих типов встречаются и в других организмах
Однако сверхспирализация ДНК имеет огромное значение не только для процессов репликации. Аккуратно выделенная из клеток ДНК находится в виде отрицательной сверхспирали. В релаксированной ДНК на один оборот двойной спирали приходится 10,5 пар оснований, а в клеточной - 12, т. е. она слабо скручена. Для ДНК, выделенной из различных клеток, степень сверхспирализации сравнима, что свидетельствует о важности и контролируемости этого явления. Известно, что у Е. coli отрицательная сверхспирализация необходима как для репликации, так и для транскрипции (см. главу 21), из чего следует, что в сверхспирализованном состоянии легче происходит отделение нитей ДНК. Также предполагают, что взаимодействие белка с отрицательно сверхспирализованными участками ДНК способствует уменьшению конформационного напряжения и увеличивает выход энергии, необходимой для связывания, иначе говоря, облегчает присоединение белков к ДНК.
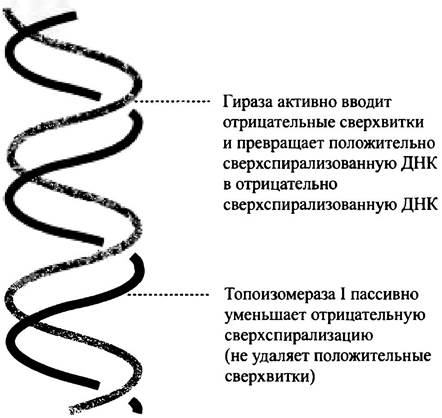
Подводя итог, следует еще раз подчеркнуть, что в клетках Е. coli необходимая отрицательная сверхспирализация ДНК создается гиразой (топоизомеразой II), а топоизомераза I релаксирует отрицательные сверхвитки, но не действует на положительные. Равновесие между двумя противоположными активностями (гиразы и топоизомеразы I) поддерживает необходимую степень сверхспирализации (рис. 20.8); правильное соотношение активностей двух ферментов необходимо для жизнедеятельности клетки. Для контроля за этим специально синтезируются два фермента; степень и знак сверхспирализации регулируется фактором, синтез которого контролируется соответствующим геном. Необходимость гиразы для жизнедеятельности бактерий подтверждается тем, что антибиотик налидиксиновая кислота угнетает их размножение путем инактивации гиразы. У эукариот этот фермент отсутствует, но налидиксиновую кислоту можно использовать для лечения определенных инфекций у человека.
Рис. 20.8. Сбалансированное действие гиразы и топоизомеразы I у Е. coli. В результате совместной работы этих ферментов достигается необходимая для репликации ДНК и транскрипции степень отрицательной сверхспирализации. Гираза - АТР-зависимый фермент. Считается, что количество и соотношение синтезированных ферментов контролируется степенью сверхспирализации промоторов генов
Интересно, что у термофильных бактерий, живущих при очень высоких температурах, обнаружена обратная гираза, механизм действия которой прямо противоположен механизму действия гиразы Е. coli. Такое действие фермента можно объяснить тем, что положительно сверх- спирализованная ДНК лучше защищена от разделения цепей, которое происходит при высоких температурах.
Эукариотическая ДНК в клетке, подобно прокариотической, недокручена или отрицательно сверхспирализована. Но, в отличие от прокариот, эукариотическая топоизомераза, которая могла бы активно вставлять отрицательные сверхвитки в ДНК, неизвестна. Что же происходит в эукариотических клетках?
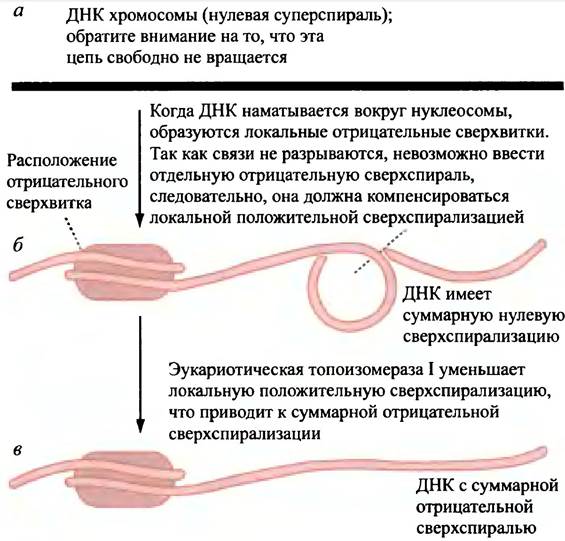
При образовании хроматина ДНК наматывается на нуклеосомную сердцевину, и в области контакта с белком молекула ДНК недокручена. Такие нуклеосомные витки не имеют разрывов, а ДНК в хромосоме не может свободно вращаться. Поэтому локальная отрицательная сверхспирализация на нуклеосоме компенсируется положительной сверхспирализацией в другом месте, так что суммарные изменения структуры сводятся к нулю (рис. 20.9, б). Эукариотические топоизомеразы релаксируют положительный сверхспиральный фрагмент за счет включения отрицательного супервитка (рис. 20.9, в). Прокариотический тип гиразы, таким образом, здесь не нужен.
Рис. 20.9 Механизм, с помощью которого эукариотическая ДНК приобретает отрицательные сверхвитки. В эукариотических клетках отсутствует фермент, способный активно включать отрицательные сверхвитки подобно прокариотической гиразе
Итак, мы имеем хеликазы, раскручивающие нити, и топоизомеразы, позволяющие разделить их благодаря уменьшению положительной сверхспирализации, вызываемой вращением двойной спирали по типу шарнирного механизма вовремя расплетания цепей. Но это еще не все: для раскручивания спирали нужны также белки SSB.
Что такое SSB?
Это белки, связывающиеся с одноцепочечной ДНК (от англ. Single Strand Binding Proteins). Они обладают большим сродством к одноцепочечной ДНК - вне зависимости от последовательности оснований - и взаимодействуют с ней по всей длине разделившихся нитей. Освобождающаяся при связывании энергия не позволяет расплетенным нитям снова соединяться; таким образом одноцепочечная форма защищается от обратимого отжига. Когда нити используются в качестве матрицы для синтеза новой ДНК, SSВ-белки удаляются.
Положение на данный момент
Пока мы имели дело с общими аспектами процесса репликации ДНК - полуконсервативным механизмом, основанном на комплементарном взаимодействии пар оснований Уотсона-Крика, биологическими аспектами клеточного цикла, инициацией репликации и механизмом раскручивания ДНК. Вышеизложенное свидетельствует о сложности процесса репликации ДНК. Мы получили представление о разделение нитей ДНК, но пока ничего не узнали о механизме синтеза ДНК в репликативной вилке. Вернемся к этой проблеме. Прежде всего надо понять: как нуклеотиды связываются между собой при синтезе новой цепи ДНК? Ферменты, катализирующие этот процесс, называются ДНК-полимеразами, так как они полимеризуют нуклеотиды с образованием молекулы ДНК.
Основные ферментативные реакции, катализируемые ДНК-полимеразами
Мы рассмотрим основную химическую реакцию образования ДНК, но прежде остановимся на некоторых аспектах.
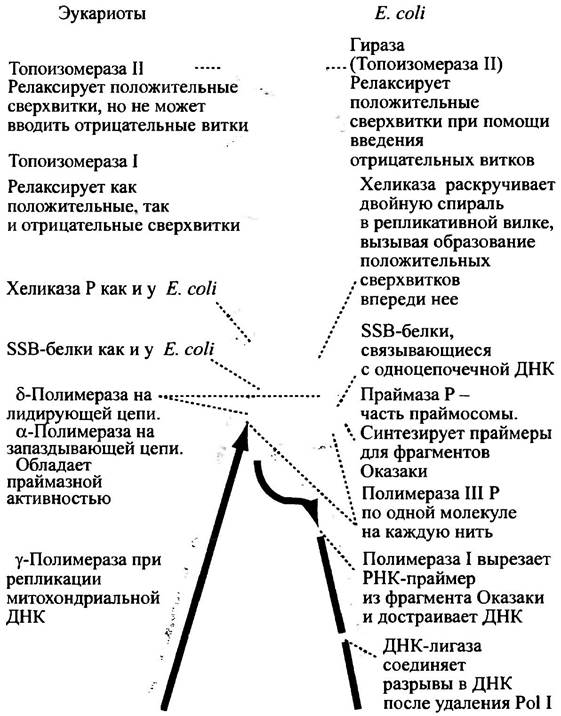
✵ У Е. coli существует три ДНК-полимеразы, называемые Pol I, Pol II и Pol III.
✵ Синтез ДНК, происходящий в репликативной вилке, катализируется Pol III или ее эукариотическими аналогами; Pol I играет важную роль как в репликации, так и в репарации ДНК. Pol II изучена меньше; предполагается, что она связана с определенным типом репарации ДНК.
✵ Субстратами ДНК-полимеразной реакции служат четыре дезоксинуклеозидтрифосфата dATP, dCTP, dGTP и dTTP. В ходе репликации образуются дочерние цепи - копии с материнских цепей.
✵ Для копирования полимеразе нужна одноцепочечная матрица ДНК. Термин копирование используется в смысле комплементарности: G на матрице «копируется» (переписывается) в С дочерней цепи; соответственно А «копируется» в Т, С - в G, Т - в А.
✵ Очень важно запомнить: ДНК-полимераза может только элонгировать (удлинять) уже существующую нить, называемую затравкой (праймером). Праймер комплементарен матрице и состоит из 2-10 нуклеотидов; без него синтез начаться не может. ДНК-полимераза не инициирует образование новой цепи, так как она не может соединить два свободных нуклеотида.
Полимераза присоединяет нуклеотиды к свободной концевой 3'-ОН-группе праймера, высвобождая при этом неорганический пирофосфат (рис. 20.10). Гидролиз фосфата увеличивает отрицательное значение ∆G°синтеза полинуклеотидной цепи, способствуя таким образом протеканию реакции полимеризации. Включение нуклеотида в новую цепь ДНК сопровождается образованием водородных связей с матрицей. При этом происходит высвобождение энергии, что делает процесс термодинамически выгодным.
Рис. 20.10. Реакция, катализируемая ДНК-полимеразой. На схеме показано присоединение аденинового дезоксинуклеотида (от АТР) к 3'-концу затравки (праймеру); выбор присоединяемого основания определяется основанием в матричной цепи. Синтез протекает в направлении 5' —> 3', т. е. цепь удлиняется в этом направлении
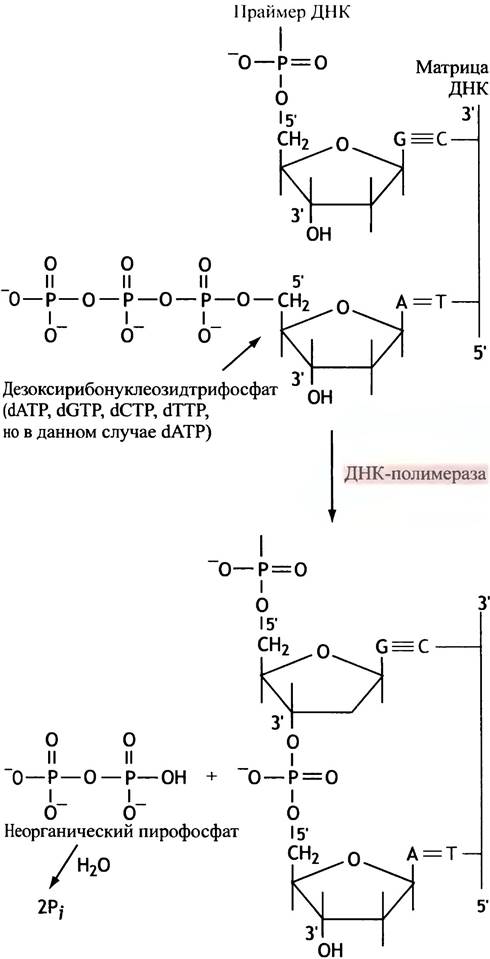
Порядок взаимодействия четырех дезоксинуклеозидтрифосфатов с ДНК-полимеразой определяют основания копируемой родительской цепи.
Синтез ДНК всегда протекает в направлении 5’ —> 3’ растущей цепи. Это означает, что растущая цепь элонгируется (наращивается) в направлении 5’ —> 3'; нуклеотид добавляется к свободному 3’-ОН-концу предшествующего нуклеотида. Напомним, что когда мы говорим о синтезе в направлении 5' —> 3', мы всегда имеем в виду направление элонгации или полярности новой нити. Матричная цепь имеет противоположную полярность. О полярности нитей ДНК мы говорили в предыдущей главе (см. с. 235).
Проблемы и еще раз проблемы при синтезе ДНК
Как инициируется новая нить?
ДНК-полимераза не может инициировать образование новой цепи, поэтому в любой начальной точке синтез должен быть инициирован.
Решение этого вопроса довольно необычно и заключается в том, что цепи ДНК инициируются при помощи РНК. РНК имеет структуру, аналогичную структуре одноцепочечной ДНК, только в качестве углеводного компонента в ней выступает рибоза, а тимин (Т) заменен на урацил (U). РНК синтезируется РНК-полимеразой практически при помощи такого же химического механизма, как и ДНК, за исключением того, что для синтеза используются рибонуклеозидтрифосфаты АТР, СТР, GTP и UTP. Но в данном случае принципиальное различие состоит в том, что РНК-полимераза способна инициировать новые цепи. Она может соединить два нуклеотида (при этом также требуется матрица), а ДНК-полимераза - нет.
После того, как специальной РНК-полимеразой, называемой праймазой, будет синтезирован небольшой участок РНК-праймера (например, из 5 нуклеотидов), подключается ДНК-полимераза и продолжает наращивать цепь.
Теперь рассмотрим антипараллельное расположение цепей в молекуле ДНК.
Как решается проблема полярности цепей при репликации ДНК?
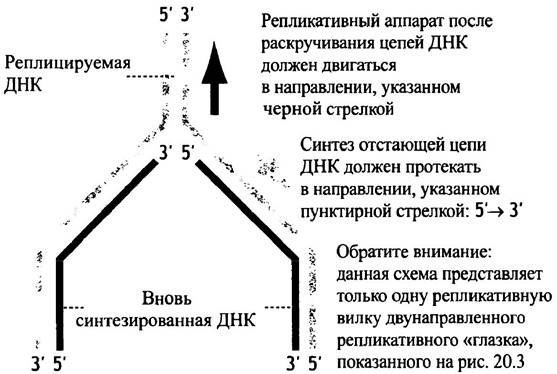
Вспомним предыдущую главу, где обсуждается полярность цепей ДНК. ДНК реплицируется в каждой репликативной вилке, которая постепенно смещается вдоль хромосомы. У Е. coli в каждой репликативной вилке работают две молекулы ДНК-полимеразы III (Pol III), по одной на цепь. Эти две молекулы объединяются в единый асимметричный димер и образуют холофермент. Молекулы полимеразы должны двигаться в одном направлении (рис. 20.11). Однако направление 5' —> 3' одной родительской цепи противоположно направлению движения вилки, а в другой цепи совпадает с ним. Поскольку ДНК-полимераза может осуществлять синтез только в направлении 5' —> 3', матричная цепь должна идти в направлении 3' —> 5', которое противоположно направлению синтеза. Это вполне подходит для синтеза одной новой нити (на рис. 20.11 - левой), а что же с другой? Она идет в направлении перемещения вилки (5' —> 3'), следовательно, синтез дочерней цепи должен идти в противоположном направлении. Рис. 20.11 отражает на первый взгляд неразрешимую проблему: полимераза на правой нити должна двигаться вверх по странице, что она и делает, а ДНК должна синтезироваться в направлении вниз по странице. Левая нить называется лидирующей цепью, другая - запаздывающей.
Рис. 20.11. Распределение полярности цепей при репликации ДНК
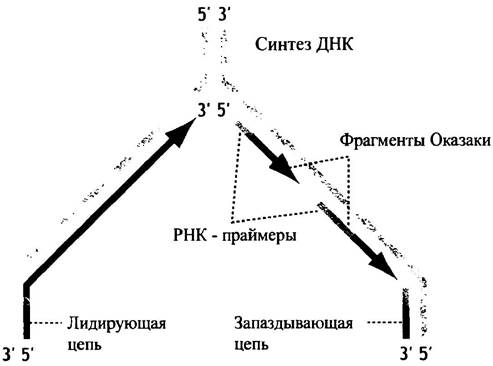
Но как же инициируется синтез запаздывающей цепи? С лидирующей цепью все ясно: праймаза закладывает простой праймер в точке начала репликации, а ДНК-полимераза наращивает цепь. Но это не подходит для запаздывающей цепи. Проблема решается следующим образом: пока цепи ДНК разъединены, на правой цепи происходит неоднократно инициированный праймазой (РНК-полимеразой) синтез множества коротких цепей ДНК (рис. 20.12). На первый взгляд это кажется невероятным!
Рис. 20.12. Репликативная вилка. Лидирующая цепь синтезируется постоянно, в то время как запаздывающая - отдельными короткими фрагментами (фрагменты Оказаки)
Короткие цепи ДНК, связанные с РНК-праймерами на запаздывающей цепи, называются фрагментами Оказаки - по имени открывшего их исследователя. Однако из схемы все еще неясно, как ДНК-полимераза может синтезировать ДНК в направлении, противоположном ее движению. Кроме того, запаздывающая цепь состоит из серии разрозненных коротких фрагментов, соединенных с праймером. Для начала разберем, как работает репликативный аппарат в репликативной вилке.
Ферментативный комплекс репликативной вилки Е. coli
Функциональный комплекс белков репликативной вилки изображен на рис. 20.13. Ключевыми ферментами являются хеликаза, раскручивающая двойную спираль, праймаза,синтезирующая праймеры на участках запаздывающей цепи, и конечно комплекс Pol III. Раскручивающая активность хеликазы зависит от АТР; фермент (вероятно, благодаря конформационным изменениям) перемещается по цепи ДНК и разделяет две цепи двойной спирали. После присоединения SSB-белков происходит стабилизация одиночных цепей. У Е. coli в репликативной вилке присутствуют две связанные между собой молекулы Pol III, одна из которых синтезирует лидирующую цепь, а другая запаздывающую. Это по сути один и тот же фермент, но димер холофермента асимметричен в связи с наличием дополнительной субъединицы со стороны запаздывающей нити. Pol III является высокопроцессивным ферментом: однажды присоединившись к цепи ДНК, он удерживается на ней на отрезке в тысячи оснований, что удобно для лидирующей цепи. Его фиксации на матричной цепи помогают так называемые кольцевые скользящие «зажимы» (β-субъединицы), состоящие из двух половинок. Они образуют позади Pol IIIскользящее кольцо, связанное с ферментом, что предотвращает отделение последнего от матричной нити ДНК. Круговой зажим имеет отверстие для свободного протягивания ДНК. Структуру зажима иллюстрирует ленточная модель (рис. 20. 14).
Рис. 20.13. Расположение ферментов в репликативной вилке Е. coli (упрощенная схема)
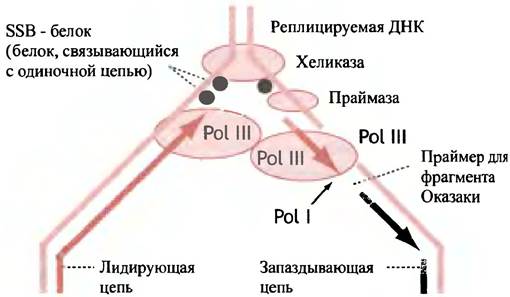
Рис. 20.14. Ленточная модель скользящих «зажимов» дрожжей и Е. coli. а - «Зажим» дрожжевых клеток, обеспечивающий процессивность ДНК-полимеразы. является тримером; б - «зажим» Е. coli, присоединяющийся к ДНК-полимеразе III, - димер. Отдельные субъединицы внутри каждого кольца показаны разными оттенками. Цепи β-слоя изображены плоской лентой, а α-спираль - спирализованной. В центре каждой структуры помещена модель В-формы ДНК, так как кольцо может охватывать дуплекс
На запаздывающей цепи фермент (вместе с кольцевым зажимом), встречая праймер, синтезированный праймазой для предыдущего фрагмента, останавливается.
Пока неизвестно, отделяется ли комплекс Pol III, чтобы присоединиться вновь после встречи с новым праймером, который будет использован для синтеза очередного фрагмента Оказаки, или все происходит как-то иначе.
Теперь перейдем к топологической проблеме, возникающей при синтезе фрагментов Оказаки в направлении 5' —> 3', так как репликативная машина не имеет обратного хода. Основной принцип предельно понятен. Если запаздывающая цепь образует петлю, то на коротком участке она будет ориентирована в том же направлении, что и лидирующая. Таким образом, репликативный аппарат может перемещаться в направлении вилки и одновременно синтезировать обе новые цепи.
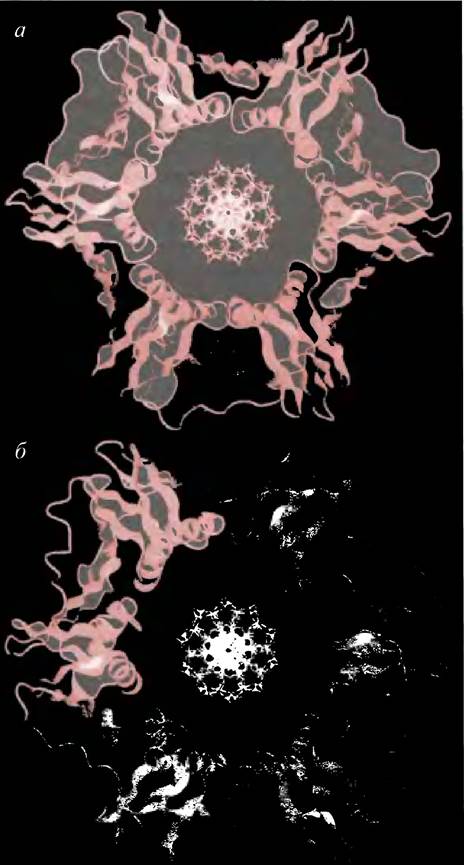
Хотя принцип прост, проблема, связанная с перемещением петли вдоль родительской цепи при синтезе фрагментов Оказаки, гораздо сложнее. Для ее решения Корнбергом была предложена модель, названная моделью петли (рис. 20.15).
Рис. 20.15. «Петельная» модель синтеза фрагментов Оказаки. Более темные стрелки указывают направление синтеза ДНК. Согласно модели, при встрече нового фрагмента Оказаки с предыдущим необходимо разрушение петли. Затем должна образоваться новая петля. В этой модели обе нити могут синтезироваться в направлении 5' —> 3', так как репликативный аппарат перемещается в направлении вилки. Точно описать детали образования петли невозможно
По мере распространения петли и перемещения репликативного аппарата новые нити ДНК будут встречать 5'-концы РНК от предыдущего фрагмента Оказаки. В этой точке полимераза должна освобождаться, петля разрушиться, а затем образоваться новая. А теперь разберемся: каким образом фрагменты Оказаки соединяются в непрерывную цепь ДНК?
Что происходит с фрагментами Оказаки?
У Е. coli Pol III отделяется от ДНК в момент достижения РНК-праймера предшествующего фрагмента Оказаки, оставляя разрыв в месте соединения ДНК и РНК. Здесь подключается ДНК-полимераза I (Pol I). Это удивительный фермент, объединяющий в одной молекуле три различных ферментативных активности.
Если мы рассмотрим схему на рис. 20.12, то увидим, что отдельные фрагменты ДНК (фрагменты Оказаки) должны быть превращены в непрерывную цепь ДНК. Каждый фрагмент начинается с РНК, которую нужно удалить, затем заделать бреши, а отдельные фрагменты ДНК соединить между собой. Pol I присоединяется к разрывам между завершенными фрагментами Оказаки, добавляет нуклеотиды к 3'-ОН-концу предыдущего фрагмента, двигаясь в направлении 5' —> 3', как и Pol III (при синтезе ДНК добавление нуклеотидов всегда происходит в направлении 5' —> 3'). По мере перемещения передняя часть Pol I сталкивается с РНК предыдущего фрагмента Оказаки и отщепляет его рибонуклеотиды, а расположенный позади участок фермента присоединяет дезоксирибонуклеотиды, заполняя бреши. «Передняя» активность называется 5' —> 3'-экзонуклеазной: экзо- потому, что фермент удаляет нуклеотиды с конца молекулы, нуклеазная - потому, что фермент гидролизует нуклеиновую кислоту, а 5' —> 3' указывает направление движения фермента от 5'-конца РНК в направлении 3'-конца. Обратите внимание на то, что Pol III, в отличие от Pol I, не обладает 5' —> 3'-экзонуклеазной активностью и при встрече с фрагментом Оказаки не может отщеплять праймер РНК. Поэтому она отделяется в этой точке от ДНК, и всю остальную работу проделывает Pol I.
Но это еще не все. «Тыльная» сторона молекулы Pol I наряду с полимеразной проявляет 3' —> 5'-экзонуклеазную активность. Эта экзонуклеаза гидролизует нуклеотиды с 3'-конца цепи ДНК, которую только что синтезировала Pol I, и отрезает их в направлении 5'-конца, т. е. в противоположном действии 5' —> 3'-экзонуклеазы.
Парадоксально, но факт: фермент, который одновременно разрывает ДНК, в тоже время синтезирует ее. Но это можно объяснить. 3' —> 5' экзонуклеаза гидролизует концевой нуклеотид вновь образованной нити ДНК только тогда, когда его основание не спаривается с соответствующим основанием матричной цепи; а ДНК-полимераза добавляет нуклеотиды только в том случае, если предыдущее основание дочерней цепи надлежащим образом спарено с соответствующим основанием матричной цепи. Так фермент проверяет правильность включения в цепь предыдущего основания, вырезает его, если оно не соответствует матрице и заменяет другим. Этот процесс называется коррекцией неправильного спаривания; с его помощью контролируется правильность синтеза.
ДНК-полимераза I (в отличие от Pol III) низкопроцессивна. Она непрочно удерживается на матричной цепи и довольно быстро отделяется от нее после удаления РНК, не имея кругового зажима, удерживающего ее на ДНК. Это понятно, иначе она продолжала бы замещение длинных цепей новосинтезированной ДНК. После ее отделения в цепи остаются разрывы, для соединения которых используется фермент, называемый ДНК-лигазой.
Этот фермент синтезирует фосфодиэфирную связь между 3'-ОН одного фрагмента ДНК и 5'-фосфатом следующего. Процесс требует затраты энергии. У некоторых прокариот и эукариот ее источником служит АТР. Механизм действия фермента (Е) состоит в том, что он принимает АМР от АТР, освобождая пирофосфат, и переносит АМР на 5'-фосфат ДНК. Затем ДНК-AMP реагирует с ДНК-3'-ОН, высвобождая АМР и заделывая разрыв:
Е + АТР —> Е-АМР + РРi;
Е-АМР + Р-5-ДНК —> Е + АМР-Р-5'-ДНК;
ДНК-3’-ОН + АМР-Р-5-ДНК —> ДНК-3’-О-Р-5'-ДНК + АМР.
АМР соединяется с ферментом и ДНК через 5'-фосфатную группу.
У Е. coli вместо АТР используется NAD+. Он может служить источником АМР точно также, как и АТР, и у Е. coli происходит именно так. Потом Pol I связывается с ДНК в определенной точке (рис. 20.16). Полимераза добавляет нуклеотиды к 3'-концу фрагмента ДНК, проверяя каждое присоединенное основание, и перемещается в направлении 5'—> 3'. РНК и некоторая часть ДНК отщепляются - и промежуток заполняется ДНК. Затем Pol I отделяется, и лигаза соединяет вместе два фрагмента ДНК. Таким образом, из серии фрагментов Оказаки образуется непрерывная новая цепь ДНК.
Рис. 20.16. Схема действия полимеразы I по преобразованию фрагментов Оказаки. dВ - Дезоксирибонуклеотид; rВ - рибонуклеотид

Коррекция неправильного спаривания оснований полимеразой III
Последовательность оснований во вновь образованной цепи ДНК должна быть воспроизведена точно, поскольку включение даже одного неправильного основания может привести к летальному исходу. Но при огромном количестве оснований ошибки неизбежны.
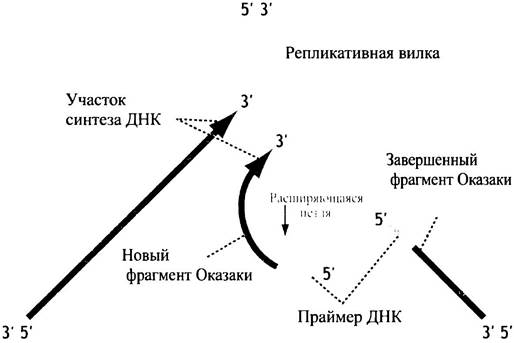
Когда Pol III встретит на матричной цепи, например, G, ее высокая специфичность проявится в том, что в абсолютном большинстве случаев только dCTP будет присоединен к активному центру фермента и перенесен на растущую цепь ДНК. Аналогичное утверждение справедливо для других оснований. Механизм, с помощью которого Pol IIIподбирает правильный нуклеотид к основанию матрицы, основан на модели Уотсона-Крика, когда все пары комплементарных оснований имеют одинаковую геометрическую форму, отличную от любой другой пары оснований; это строго распознается ферментом (рис. 20.17). Образование водородных связей между нужными парами и между основаниями одной цепи (стэкинг-взаимодействия) также способствуют правильному отбору, поскольку это сопряжено с высвобождением энергии.
Рис. 20.17. Геометрические характеристики пар оснований Уотсона-Крика и «ошибочных» пар. Данные получены на основе рентгенографии кристаллов двунитевых олигодезоксирибонуклеотидов (В-форма ДНК). Наблюдается строгое геометрическое соответствие пар Уотсона-Крика и неустойчивое состояние «качающихся» пар А-С и G-Т или пары G-А (в конформации анти- и син- соответственно). Термин «качающиеся» пары будет объяснен на с. 287. Указаны расстояния (в нм) между С-1-атомами дезоксирибозы и углы вращения гликозидной связи между основанием и дезоксирибозой
Впрочем, этот механизм имеет предел точности. Хотя образование пар по Уотсону-Крику является определяющим моментом, в любом наборе нуклеотидов несколько молекул, например, dATP, будут находиться в имино-форме (=NH вместо -NH2), и при этом они могут спариваться с С. Следовательно, Pol III может совершать ошибки при копировании ДНК с частотой примерно одну на 105-106 оснований. Это дало бы слишком высокий уровень мутаций. Однако у Е. coli реальная конечная частота ошибок при репликации ДНК значительно ниже, около 10-10 за один раунд репликации.
Оказалось, что Pol III обладает также 3' —> 5'-экзо- нуклеазной активностью, благодаря которой последнее из включенных в синтезируемую цепь оснований может быть удалено, если оно неправильно спаривается. В результате такого устранения ошибок считывания их уровень сводится к минимуму. Большинство событий, происходящих при репликации ДНК у Е. coli и эукариот, показано на рис. 20.18.
Рис. 20.18. Репликации ДНК Е. coli и эукариот
Метил-зависимое исправление (репарация) ошибок
Несмотря на использование описанных выше механизмов устранения ошибок считывания, обеспечивающих высокую точность в системе репликации ДНК, ошибки при работе с таким огромным числом присоединяемых нуклеотидов, неизбежны. У Е. coli имеется другой механизм; он используется для исправления ошибок в последовательности оснований вновь синтезированной ДНК. Если ошибка не была устранена полимеразой, она вызовет деформацию дуплекса.
Ошибка появляется в новой цепи, и репарирующая система обязана распознать именно ее. Важно, что при ошибках такого рода основание родительской цепи является правильным по определению, а неправильным является комплементарное основание в новой цепи. Репарирующая система должна четко различать обе цепи
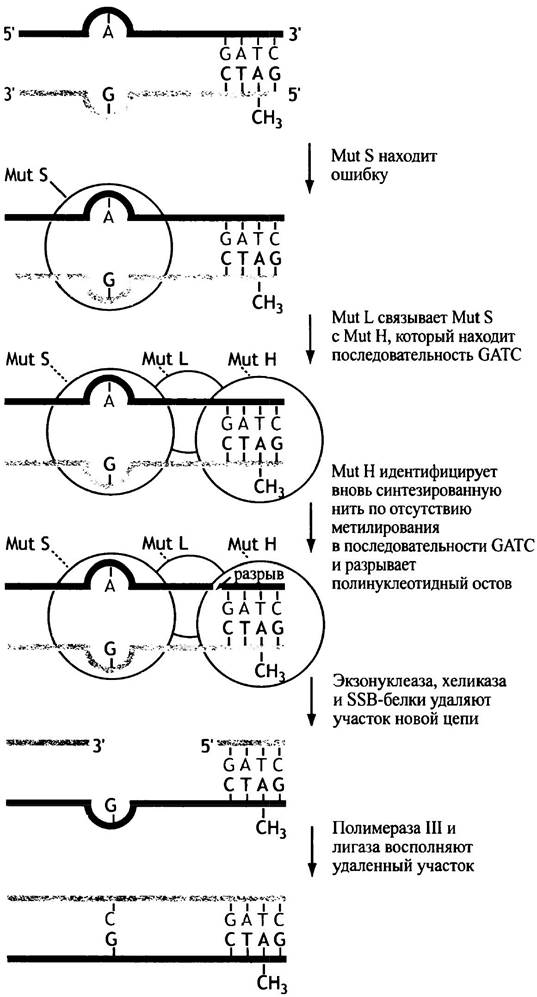
(родительскую и дочернюю), чтобы удалить мутированное основание из новосинтезированной цепи и исправить ошибку. Как же происходит распознавание цепей? В ДНК Е. coli в последовательности GATC аденин всегда метилирован ферментом Dam-метилазой. Метилирование основания не нарушает спаривания оснований или структуры ДНК, так как оно происходит спустя некоторое время после синтеза новой цепи. Таким образом, в течение какого-то времени родительская нить будет метилирована, а вновь синтезированная - нет. Репарирующая система включает меньше компонентов по сравнению с системой, участвующей в репликации ДНК. Один белок Mut S узнает нарушение в спирали; Mut Н, связанный с Mut S при участии Mut L, находит неметилированную последовательность GATC на одной из цепей дуплекса (идентифицируя таким образом вновь синтезированную цепь) и разрывает ее в этом месте (рис. 20.19). Место разрыва может быть удалено от места ошибки на тысячи оснований. Затем, при совместном действии хеликазы, SSB-белков и экзонуклеазы, удаляется участок цепи от разрыва до места ошибки, а полимераза III застраивает образовавшуюся брешь в ДНК. Завершает репарацию ДНК-лигаза, соединяющая нуклеотиды в месте разрыва. Для исправления одного основания бывает необходимой замена тысячи оснований (см. рис. 20.19). Эта система исправления ошибок тысячекратно увеличивает точность репликации. Подобная система репарации ошибок существует и у эукариот. У человека известны белки, аналогичные белкам Mut S и Mut L Е. coli. Мутации в их генах сопровождаются увеличением риска возникновения раковых опухолей.
Рис. 20.19. Метил-зависимый путь репарации ошибок
Репарация повреждений ДНК у Е. coli
Описанный выше механизм обеспечивает репликацию ДНК с очень высокой степенью точности, гарантирующей продолжение жизни. Однако все еще остается большая проблема. Последовательность оснований в ДНК обязательно должна быть неизменной, поскольку информация, которую она несет, необходима организмам в течение всей их жизни. Но ДНК - с точки зрения химической стабильности - обычная молекула, и с ней могут происходить химические изменения, которые способны вызывать большое число мутаций, если бы не процессы постоянной репарации. Некоторые повреждения носят спонтанный характер. Гликозидная связь, при помощи которой пуриновые и (в меньшей степени) пиримидиновые основания соединяются с остатками дезоксирибозы, нестабильна, и нередко происходит апуринизация и апиримидинизация, когда большое количество пуринов и пиримидинов отщепляется от ДНК. Кроме этого, цитозин и аденин подвергаются дезаминированию, превращаясь в урацил и гипоксантин соответственно (см. формулы на с. 217 и рис. 18.7). ДНК также является объектом атаки множества соединений.
Ионизирующая радиация, свободные радикалы кислорода (см. с. 212), канцерогены, УФ-излучение могут повреждать ДНК и, следовательно, вызывать мутации.
В общем, повреждения, затрагивающие обе нити двуспиральной молекулы ДНК и происходящие точно в одном и том же месте обеих цепей, относительно редки.
хотя и случаются. При повреждении какого-то участка одной из цепей вторая цепь всегда может служить матрицей и управлять репарацией поврежденной части. В клетке существует множество систем, репарирующих практически любые повреждения ДНК, поскольку поддержание целостности этой молекулы имеет исключительное биологическое значение. ДНК нужно репарировать любой ценой.
Существуют различные типы репарирующих систем.
✵ Прямая репарация. Облучение ДНК ультрафиолетовыми лучами может вызывать ковалентное связывание двух расположенных рядом (в одной и той же цепи) тиминовых оснований с образованием Т-димера.
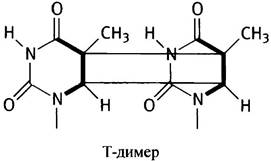
У Е. coli нехарактерные связи разрываются при помощи активируемого светом механизма, благодаря чему остатки тимина возвращаются в исходное состояние. Система широко распространена и имеет большое значение для растений. Алкильные группы оснований (они- образуются под действием некоторых мутагенов) могут напрямую удаляться под действием так называемого «фермента-самоубийцы»: он служит их акцептором, после чего сам подвергается разрушению. Строго говоря, это скорее специфичный белковый реагент, чем фермент.
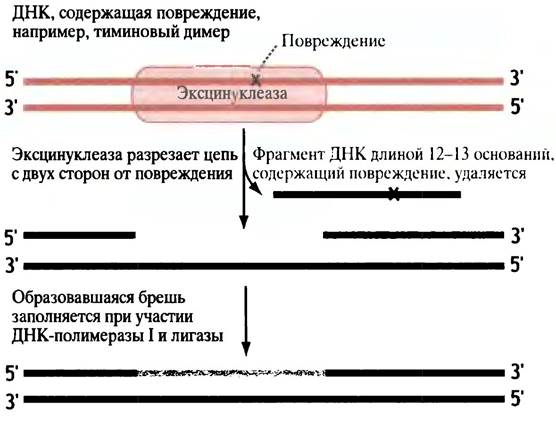
✵ Репарация с вырезанием (эксцизией) нуклеотида. Повреждения, деформирующие двойную спираль, например, Т-димеры, могут также репарироваться путем вырезания включающих повреждение коротких последовательностей нуклеотидов с последующим замещением их на правильную последовательность. При этом в качестве матрицы используется противоположная цепь ДНК. У Е. coli есть необычная эндонуклеаза: эксцинуклеаза, или комплексuvrABC - по названию трех генов, кодирующих фермент. Она разрезает ДНК по обе стороны от повреждения и удаляет однонитевой фрагмент из 12-13 нуклеотидов (рис. 20.20). К разрыву присоединяется ДНК-полимераза I и добавляет нуклеотиды к 3'-концу разорванной цепи; лигаза соединяет разрыв. Работа этой системы зависит от правильного распознавания поврежденной нити ДНК.
Рис. 20.20. Путь эксцизионной репарации у Е. coli
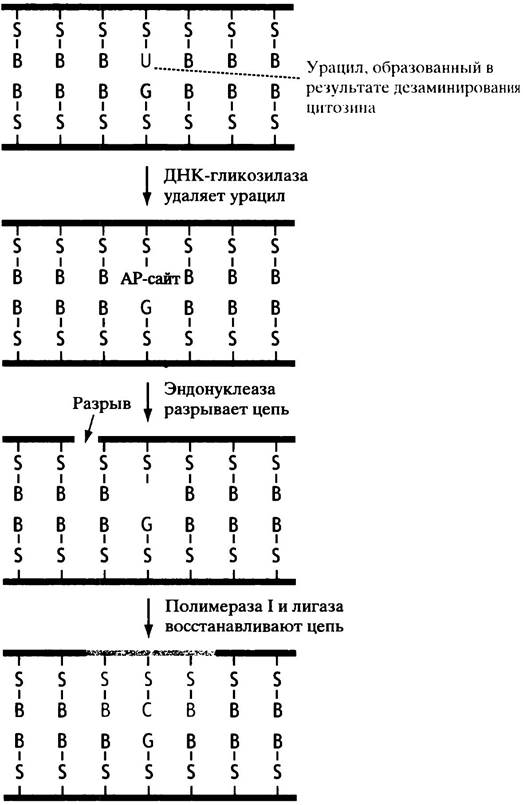
✵ Репарация с эксцизией основания и репарация АР-сайта. Дезаминирование превращает цитозин в урацил и аденин - в гипоксантин. ДНК-гликозидаза узнает обычное основание и отщепляет его, оставляя АР-сайт (от англ. apurinic-apyrimidinic) - дезоксирибозу, лишенную основания (рис. 20.21). АР-сайты могут также образовываться спонтанно, поскольку связь пурин-дезоксирибоза достаточно нестабильна. Репарация АР-сайтов включает разрезание полинуклеотидной цепи, прилежащей к месту повреждения, с последующим замещением этого фрагмента при участии ДНК-полимеразы I и лигазы. Образованный из цитозина урацил также необходимо удалять (вероятно, это и объясняет, почему в состав ДНК вместо и входит Т). Напомним, что Т отличается от и наличием дополнительной метильной группы (см. с. 217). Если бы нормальная ДНК содержала и, то было бы невозможно отличить обычный и от «ложного», образованного при дезаминировании С.
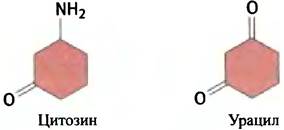
Рис. 20.21 Образование и репарация АР-сайта: S - сахар; В — основание. В приведенном примере АР-сайт образуется после удаления урацила под действием гликозилазы. Но такие сайты могут возникать и в ходе спонтанного гидролиза пуриновых оснований (в меньшей степени - пиримидиновых оснований)
Использование в составе ДНК основания Т вместо U решает эту проблему. Как будет рассказано в следующей главе, U используется в составе РНК, но поскольку она имеет относительно более короткое время жизни и гораздо меньшие размеры, ошибки в ней не так критичны. Вероятно, поэтому РНК и не репарируется. Замечена довольно четкая корреляция: в клетках Е. coli может присутствовать множество разрывов ДНК, требующих присоединения Pol I и проведения ею репарации, но имеется только две репликативных вилки, требующие участия Pol III. Поэтому в клетках Е. coli присутствует всего 10-20 молекул Pol III и значительно больше молекул Pol I.
Устройство репликативной вилки эукариот
Общие принципы репликации ДНК у Е. coli применимы и к эукариотам, правда, с некоторыми поправками. Главная заключается в том, что вместо димера Pol III у Е. coli
в синтезе лидирующей и запаздывающей цепей участвуют разные полимеразы.
У эукариот описано пять ДНК-полимераз, которые обозначаются греческими буквами. Полимеразы α и δ участвуют в репликации ядерной ДНК (Pol III у Е. coli). Pol α находится в комплексе с праймазой, но не проявляет активности 3' —> 5'-экзонуклеазы. Полимераза δ обладает 3' —> 5' экзонуклеазной активностью. Похоже, что полимеразы α и δ формируют у эукариот репликативный комплекс для синтеза лидирующей цепи. δ-Полимераза дрожжей соединена с круговым скользящим зажимом, известным как PCNA (от англ. Proliferating Cell Nuclear Antigen - ядерный антиген пролиферирующих клеток) и обеспечивающим ей процессивность, как и у фермента Е. coli. На рис. 20.14 на примере ленточной модели показана структура зажимов дрожжей и Е. coli. Несмотря на внешнее сходство, их аминокислотные последовательности сильно различаются; кроме того, зажим Е. coli является димером, а дрожжевой белок - тример. Это свидетельствует о независимом формировании этих белков в ходе эволюции.
Две другие ДНК-полимеразы эукариот - β и ε - выполняют репарирующую функцию, а у-полимераза участвует в репликации митохондриальной ДНК.
Проблема репликации концов хромосом эукариот
Хромосомы эукариот линейны, что вызывает проблемы, не возникающая при репликации кольцевых хромосом.
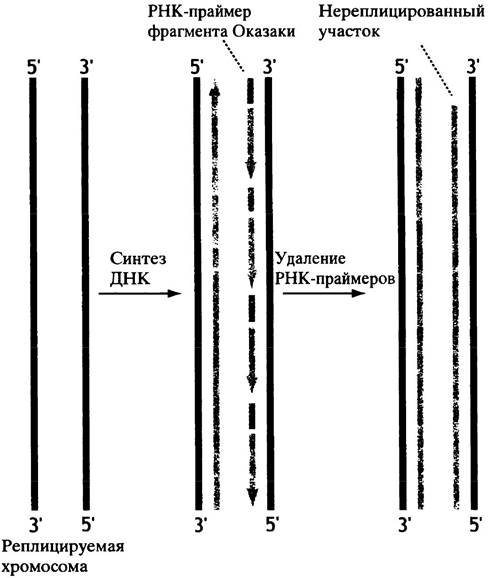
Рассмотрим репликацию хромосомы, представленной на рис. 20.22. Она реплицируется в двух направлениях от одного инициирующего сайта, расположенного в центре (на самом деле хромосома эукариот имеет много таких сайтов). 5'-концевая область лидирующей цепи реплицируется полностью. Но этого не происходит при синтезе запаздывающей цепи, потому что участвующий в образовании фрагментов Оказаки РНК-праймер расположен у 3'-конца матричной цепи (см. рис. 20.22). После удаления праймера эти концы оказываются нереплицированными, и нет механизма, позволяющего восполнить их, используя ДНК-синтезирующий комплекс, описанный выше. Для восполнения теряемых частей требуются РНК-праймеры, но матриц, с которых их можно было бы образовать, тоже нет. Значит, с каждым клеточным делением хромосомы будут последовательно укорачиваться; дальнейшие предположения затруднительны, так как механизм синтеза ДНК подразумевает полную репликацию двуцепочечной ДНК. Решение проблемы состоит в том, что на концах хромосом эукариот находятся специальные повторяющиеся последовательности ДНК - теломерная ДНК (содержащие ее концы хромосом - теломеры). Концы все еще не могут быть реплицированы при помощи описанного ранее синтезирующего ДНК комплекса, но полнота репликации «настоящей» хромосомы осуществляется при помощи праймера на 3'-конце матричной цепи, расположенной против теломерной ДНК. Более того, в быстроделящихся клетках теломера добавляется, чтобы хромосома никогда не подвергалась риску укорочения.
Рис. 20.22. Схематичное изображение укорочения вновь образованных линейных хромосом при репликации. Представлена двунаправленная репликация очень короткого фрагмента ДНК. Следует отметить, что удаление праймеров из фрагментов Оказаки происходит постоянно (здесь оно представлено как отдельное событие). У обычной хромосомы имеется множество точек начала репликации. Светлым выделены синтезируемая ДНК и РНК-праймеры фрагментов Оказаки
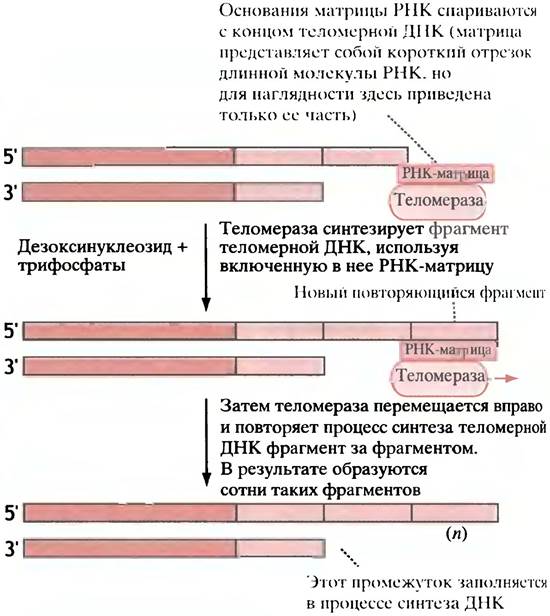
Как синтезируется теломерная ДНК?
Теломера состоит из коротких повторяющихся фрагментов (последовательность расположения оснований в этих фрагментах варьирует у разных видов). У человека имеются сотни тандемных повторов последовательности ТТАGGG. Фермент теломераза добавляет эти последовательности (одну за другой) к 3'-концу предсуществующей теломерной ДНК и таким образом удлиняет ее. Теломераза имеет две особенности: I) использует РНК в качестве матрицы для синтеза ДНК - это обратная транскриптаза (см. главу 23); 2) эта матрица включена в структуру фермента. У человека РНК имеет последовательность, включающую 1,5 повтора комплементарных ТТАGGG. Матрица РНК гибридизуется с концом теломерной ДНК (рис. 20.23), затем постепенно (по одному нуклеотиду) добавляется еще один фрагмент. После этого фермент перемещается и связывается с концом нового, только что образовавшегося фрагмента. Таким образом, теломера непрерывно достраивается. На удлиненной теломере одной цепи затем формируется комплементарная цепь, так что теломера имеет двуцепочечную структуру. Однако механизм образования такой структуры пока не выяснен. По-видимому, в этом процессе участвует ДНК-полимераза.
Рис. 20.23. Механизм синтеза теломеразой теломерной ДНК. Темными линиями обозначена хромосомная ДНК («настоящая» хромосома), а светлыми - предсуществующая на одном конце хромосомы теломерная ДНК. В теломеразу встроена короткая молекула РНК, содержащая последовательность, комплементарную повторяющемуся видоспецифичному фрагменту. Фермент прикрепляется за счет взаимодействия РНК с расположенными на конце основаниями предсуществующей теломеры и добавляет (последовательно по одному основанию) один повторяющийся фрагмент. Синтез всегда идет в направлении 5' —> 3'. Фермент перемещается таким образом, что матрица РНК постоянно спарена с концевыми основаниями нового повторяющегося фрагмента, затем добавляется следующий фрагмент и так далее. Заново синтезированная теломерная ДНК служит матрицей для построения противоположной цепи, так что теломера присутствует в обеих цепях
Необходимость теломер доказана на примере YАСs, или искусственных дрожжевых линейных хромосом. Они включают три участка ДНК, необходимых для репликации хромосомы: центромеры, точки начала репликации и теломеры. Показано, что после встраивания в дрожжевые клетки YАСs могут сохраняться в течение нескольких поколений, но по мере потери теломерных концов они со временем исчезают.
Этого не происходит у прокариот, поскольку в кольцевой хромосоме всегда есть матрица, доступная для образования праймера. Существование теломер у эукариот очень важно. Теломераза обнаружена в эмбриональных клетках, которые постоянно и быстро делятся, и непрерывное удлинение хромосом необходимо для компенсации их постоянного укорочения.
Однако в соматических клетках, которые делятся не часто, добавление теломер не столь важно. И в самом деле, теломераза в них не обнаружена. Если это так, то напрашивается следующий вывод: соматические клетки получают свою «порцию» теломерной ДНК, необходимую на время жизни клетки и ее потомства. Теломеры в большинстве клеток укорачиваются с возрастом, и это может являться важным фактором, определяющим продолжительность жизни. Если соматические клетки трансформируются в канцерогенные, они переходят в состояние неограниченного деления, в результате чего становятся бессмертными - нет предела их способности к делению в культуре. Интересно, что в раковых клетках доказано присутствие теломеразы.
Репарация повреждений ДНК у эукариот
Деалкилирующий «суицидный» фермент, описанный для Е. соli, обнаружен и в клетках человека. У эукариот также имеет место эксцизионная репарация. Например, у людей с генетическим заболеванием - ксеродермальным пигментозом - эксцизионная репарация пиримидиновых димеров обычно не встречается. При УФ-воздействии на кожу в результате ковалентного связывания двух соседних тиминовых оснований (одной цепи) образуется тиминовый димер. Пребывание пациентов с ксеродермальным пигментозом на солнце вызывает рак кожи. Таким образом, эксцизионная репарация имеет большое значение для человека. Описаны гены белков, соответствующих Mut S и Mut L Е. coli (см. с. 254); установлено, что мутации в этих генах связаны с развитием рака.
Системы репарации у эукариот играют большую роль в сохранении целостности генома. Сегодня многие исследователи выясняют связь старения человека и снижения способности к репарации ДНК, в том числе с увеличением частоты мутаций.
Транспозоны, или прыгающие гены
Оказывается, некоторые фрагменты ДНК могут перемещаться с одного места на другое в пределах одной хромосомы или встраиваться в другую хромосому. Существование прыгающих генов впервые было показано Б. Мак-Клинток при изучении генетики кукурузы. Она выяснила, что элементы регуляторного гена перемещаются в геноме с одного места на другое и влияют на экспрессию генов, приводя к появлению фенотипических вариаций. Эта работа не привлекала внимания в течение 30 лет, пока исследования на Е. coli не подтвердили способность генов к передвижению и внедрению в другое место генома, и Мак-Клинток была присуждена Нобелевская премия. Способные к перемещению последовательности ДНК получили название транспозонов, или прыгающих генов.
Простейший тип транспозона Е. coli называется инсерционной последовательностью (англ. insertion sequence, IS). Он представляет собой отрезок ДНК, включающий ген транспозазы - фермента, ответственного за перемещение. На первый взгляд, присутствие IS вызывает недоумение: ген, не кодирующий ничего, кроме возможности перемещения с места на место! IS может встроиться в любой участок хромосомы, поскольку для него не существует специфичности встраивания в определенные районы хромосомы. Когда транспозон встраивается в какой-то ген, то последний инактивируется, так как в нем нарушается нуклеотидная последовательность; однако частота перемещений довольно низка. Кроме гена транспозазы инсерционная последовательность содержит на концах инвертированные концевые повторы. Они состоят из коротких фрагментов, ориентированных как показано на рис. 20.24.
Рис. 20.24. Встраивание транспозона (типа IS) в хромосому. Точки разрывов в последовательности ДНК-реципиента показаны стрелками
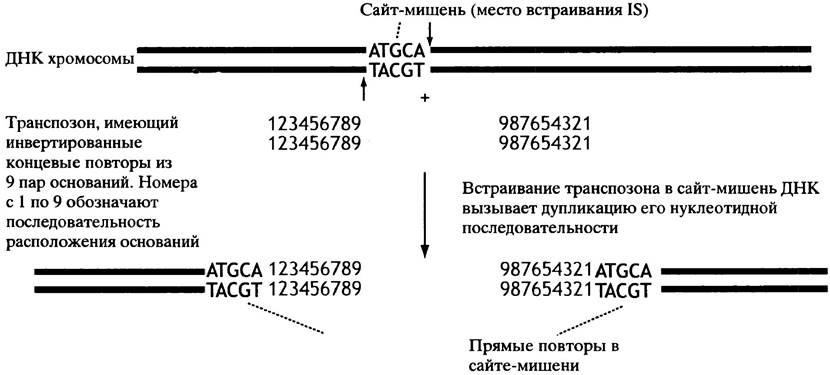
Возможны варианты механизмов транспозиций. Один из них - предварительная репликация транспозона, а не его перенос. Копия транспозона, покинув свое исходное положение, встраивается в какое-то другое место. Появление множества повторяющихся последовательностей в геноме - это путь создания благоприятных возможностей для гомологичной рекомбинации. Возможно, что перегруппировки в хромосомах, осуществляемые при участии транспозонов, чрезвычайно важны для эволюционного процесса.
Является ли единственным описанный механизм, при помощи которого синтезируется ДНК?
Известно, что для достижения одних и тех же целей эволюция использовала различные пути. Например, для синтеза цепей ДНК некоторые вирусы используют в качестве промотора белки вместо РНК. Однако ретровирусы имеют принципиально другой механизм синтеза - у них РНК генома копируется в ДНК (см. главу 23). Интересно, что митохондрии имеют собственную ДНК, которая должна реплицироваться при увеличении их численности. В то же время в митохондриях нет систем репарации ДНК. Предположительно это связано с тем, что их хромосома значительно короче, и поэтому снижается вероятность появления ошибок при репликации; а так как в клетке содержится большое число митохондрий, незначительное количество дефектов в них не столь существенно. Однако отсутствие репарации означает, что мутации в митохондриальной ДНК могут со временем накапливаться и, возможно, обусловливать развитие процессов старения. Известно большое количество генетических заболеваний, связанных с мутациями митохондриальных ДНК.
Вопросы к главе 20
1. Что такое репликон?
2. Какие топологические проблемы возникают при разделении нитей родительской ДНК в ходе репликации?
3. Как разрешаются проблемы, обозначенные в предыдущем вопросе, у Е. coli и у эукариот?
4. Объясните при помощи схем действие топоизомераз I и II.
5. У эукариот нет топоизомеразы, способной вводить отрицательные сверхвитки в ДНК. В то же время у эукариотической ДНК такие сверхвитки есть. Объясните, как они образуются.
6. Что является субстратом для синтеза ДНК?
7. Почему при синтезе ДНК используется ТТР, а не UTP, как в РНК?
8. А. Может ли цепь ДНК синтезироваться с использованием только четырех нуклеозидтрифосфатов? Объясните свой ответ. Б. В каком направлении происходит синтез ДНК? Объясните свой ответ.
9. Какие термодинамические силы движут синтезом ДНК?
10. Полимераза III Е. соli является высокопроцессивной. Объясните, что это значит, за счет чего достигается высокая процессивность и причины ее возникновения.
11. Полимераза I Е. coli является сложным ферментом. Какой активностью обладают его субъединицы и какова их роль в синтезе ДНК?
12. При помощи какого механизма ДНК-полимераза III Е. coli достигает высокой степени точности при синтезе ДНК?
13. Для достижения требуемого уровня точности считывания активности полимеразы III Е. coli недостаточно. При встраивании неправильного нуклеотида его необходимо заменить. Следовательно, репарирующая система должна распознавать ошибочное основание. Как это происходит? Существует ли такой механизм в клетках человека?
14. Объясните, что такое тиминовый димер, как он образуется и как репарируется.
15. Объясните, как укорачиваются хромосомы эукариот при каждой последующей репликации.
16. Объясните, почему предотвращается укорочение ДНК.