Основы биохимии Том 3 - А. Ленинджер 1985
Молекулярные механизмы передачи генетической информации
Синтез белка и его регуляция
Вирусные ДНК иногда содержат гены внутри других генов или перекрывающиеся гены
Долгое время основополагающим принципом молекулярной биологии считался тот факт, что нуклеотидная последовательность гена в точности коллинеарна последовательности транскрибированной с него мРНК и далее последовательности кодируемого им полипептида. Однако мы видели, что многие эукариотические гены содержат вставочные нетранслируемые нуклеотидные последовательности - интроны, которые нарушают абсолютную коллинеарность гена и кодируемого им полипептида (разд. 27.28 и 28.23).
Дополнительные вопросы относительно принципа коллинеарности возникли в результате другого удивительного открытия. Обнаружилось, что в ряде вирусов одна и та же нуклеотидная последовательность ДНК кодирует два различных белка, для чего используются две разные рамки считывания кодонов. Существование таких “генов внутри генов” было показано при изучении ДНК бактериофага фХ174, которая состоит, как выяснилось, из 5386 нуклеотидных остатков, а этого недостаточно, чтобы кодировать девять разных белков, синтезируемых этим вирусом. Как только Сэнгер и его коллеги установили полную нуклеотидную последовательность хромосомы фХ174 (с. 850), они внимательно проанализировали ее и сопоставили с аминокислотной последовательностью белков, кодируемых генами фХ174. В результате в ДНК фХ174 было обнаружено несколько перекрывающихся генных последовательностей. На рис. 29-23 показано, что гены В и Е расположены внутри соответственно генов А и D и что в пяти случаях стартовый кодон одного гена перекрывает терминирующий кодон другого. Рис. 29-24 поясняет, каким образом гены D и Е занимают один и тот же участок ДНК, но используют разные рамки считывания; аналогичная ситуация имеет место в случае генов А и В. Протяженность перекрывающихся последовательностей и внутренних генов полностью компенсирует нехватку нуклеотидов в ДНК фХ174, если учесть общее число аминокислотных остатков, содержащихся в девяти кодируемых ею белках.
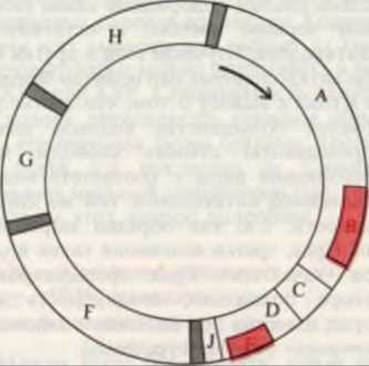
Рис. 29-23. Гены внутри генов. ДНК фага фХ174 содержит девять генов (А-J). Белки, кодируемые этой ДНК, содержат большее число аминокислотных остатков, чем может быть закодировано 5386 нуклеотидами, найденными в ДНК фага. Поэтому было высказано предположение, согласно которому какая-то часть ДНК должна кодировать больше чем один ген. При сравнении нуклеотидной последовательности ДНК с аминокислотными последовательностями кодируемых ею белков обнаружилось, что ген В расположен внутри гена А, но для этих двух генов используются разные рамки считывания. Аналогичным образом, ген Е лежит внутри гена D и также имеет другую рамку считывания (см. рис. 29-24). “Внутренние” гены обозначены красным цветом; серым цветом показаны нетранслируемые спейсерные участки ДНК. Кроме генов В и Е, расположенных внутри генов А и D, в ДНК фага фХ174 имеются короткие участки перекрывания между другими генами. В пяти случаях инициирующий сигнал одного гена перекрывает терминирующий сигнал предыдущего; при этом сигналы используют разные рамки считывания.
За этим открытием вскоре последовали подобные наблюдения и на других вирусных ДНК, в том числе на ДНК фага λ, онкогенного обезьяньего вируса 40(SV40) и фага G4-близкого родственника фага фХ174. Фаг G4 замечателен тем, что у него есть по крайней мере один кодон, который используется тремя различными генами. Было высказано предположение, что перекрывающиеся гены или гены внутри генов существуют только в геномах вирусов. Фиксированный размер вирусного капсида обусловливает экономное использование ограниченного количества ДНК, которая должна кодировать белки, нужные для обеспечения инфекционности вирусной частицы и для поддержания ее способности к репликации.
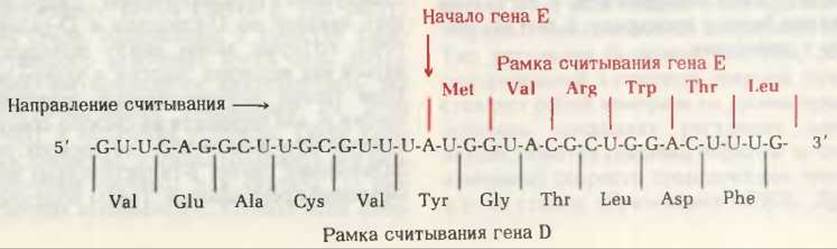
Рис. 29-24. Участок нуклеотидной последовательности транскрипта мРНК гена D в ДНК фХ174. Видно, каким образом ген Е, лежащий внутри гена D, кодируется рамкой считывания, отличной от рамки считывания гена D. Рамки считывания генов обозначены разным цветом.