Молекулярная биология клетки - Том 2 - Албертс Б., Брей Д., Льюис Дж., Рэфф М., Робертс К., Уотсон Дж. 1993
Внутриклеточная сортировка макромолекул и сохранение клеточных компартментов
Синтез и процессинг РНК
До сих пор мы говорили о хромосоме, как об очень крупном комплексе ДНК и белка, который перед делением клетки реплицируется. Однако главное предназначение хромосомы - служить матрицей для синтеза молекул РНК, поскольку только в таком виде генетическая информация, заключенная в хромосомах, непосредственно используется клеткой. РНК синтезируется весьма интенсивно: общая скорость включения нуклеотидов в РНК во время интерфазы примерно в 20 раз превышает скорость включения нуклеотидов в ДНК в S-фазе.
Синтез РНК (транскрипция ДНК) обладает высокой специфичностью. Например, в большинстве клеток млекопитающих функциональные последовательности РНК копируются приблизительно с 1% последовательностей ДНК (зрелая матричная РНК или структурная РНК). Отбор происходит на двух уровнях, которые будут по очереди обсуждаться в этом разделе: 1) для образования ядерных РНК транскрибируется лишь часть последовательности ДНК и 2) только меньшая часть нуклеотидных последовательностей ядерной РНК остается после процессинга, который предшествует экспорту молекул РНК в цитоплазму. Начнем с описания РНК-полимеразы - фермента, катализирующего всю транскрипцию ДНК.
9.4.1. РНК-полимераза, начиная каждую новую цепь РНК, меняет субъединицы [42]
Общее описание транскрипции ДНК дано в гл. 5. Транскрипция начинается, когда молекула РНК-полимеразы связывается с последовательностью промотора в ДНК. В ходе инициации две цепи ДНК локально расходятся, образуя открытый комплекс, в котором матричная цепь оказывается экспонированной. После образования этого комплекса полимераза начинает движение вдоль ДНК, удлиняя растущую цепь РНК в направлении от 5' к 3' путем последовательного добавления рибонуклеозидтрифосфатов. Так происходит до тех пор, пока не будет достигнут сигнал остановки (терминации), где вновь синтезированная цепь РНК и полимераза отсоединяются от ДНК, Таким образом, каждая молекула РНК - это одноцепочечная копия нуклеотидной последовательности ДНК, представляющей относительно короткий участок генома (см. рис. 5-1).
РНК-полимеразы обычно состоят из нескольких полипептидных цепей и имеют мол. массу 500000 дальтон и более. У бактерий и эукариот эти ферменты эволюционно близки (рис. 9-64). В связи с тем, что бактериальный фермент изучать гораздо легче, его свойства послужили основой для понимания того, как функционирует аналогичный фермент эукариот. РНК-полимераза E.coli содержит пять субъединиц: а, ß, ß', σ и ω, (по две копии а и по одной каждой из субъединиц). По нуклеотидной последовательности гена реконструирована полная аминокислотная последовательность каждой субъединицы, а данные футпринтинга ДНК свидетельствуют о том, что при соединении с ДНК фермент закрывает 60 нуклеотидных пар.
Сигма (а)-субъединица РНК-полимеразы E.coli имеет специфическую функцию и служит фактором инициации транскрипции. Эта субъединица дает возможность ферменту найти консенсусные последовательности промотора. РНК-полимеразы, узнающие разные промоторы, содержат различные формы а -субъединицы. Связавшись с промотором, фермент, для того чтобы начать синтез РНК, вступает в ряд реакций (рис. 9-65). После того, как синтезировано примерно восемь нуклеотидов молекулы РНК (стадия 4, рис. 9-65), а -субъединица диссоциирует, а вместо нее с молекулой фермента соединяется несколько факторов элонгации, необходимых для удлинения и терминации цепи. К факторам элонгации относится несколько белков, свойства которых хорошо изучены, хотя еще не совсем ясно, как они действуют.
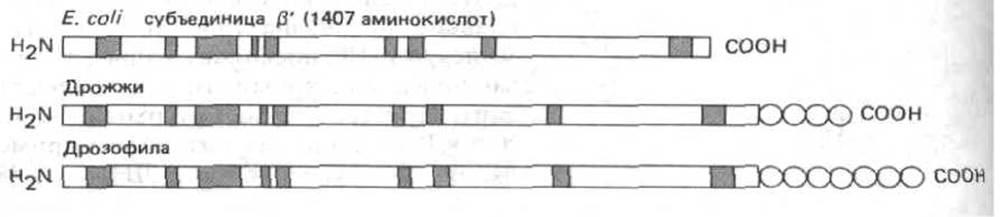
Рис. 9-64. Сходство аминокислотной последовательности бактериальных и эукариотических РНК-полимераз дает основание предположить, что эти ферменты имеют общее эволюционное происхождение. Полагают, что с ДНК связываются самые большие субъединицы РНК-полимераз из клеток Е. coli, дрожжей и дрозофилы (они сравниваются на рисунке). Закрашенные участки последовательности гомологичны более, чем на 70% у дрожжей и дрозофилы и более, чем на 40% - у дрозофилы и Е. coli. Показано, что на карбоксильном конце эукариотических субъединиц имеется уникальная последовательность Ser-Pro-Ser Туг-Scr-Pro-Thr, функция которой неизвестна: у дрожжей она повторяется 26 раз, а у лрозофилы-более 40 раз (обозначено кружками), (По A. Z. Green-leaf et al., In: RNA Polymerase and the Regulation of Transcription [W.S. Reznikoff et al., eds.] pp. 459-464. New York: Elsevier, 1987.)

Рис. 9-65. Условная схема инициации синтеза РНК, катализируемого РНК-полимеразой. Указанные стадии выявлены при изучении фермента у Е. coli. Показана молекула ДНК, несущая последовательность промотора для полимеразы Е. coli (см. рис. 5-6). Вначале фермент образует закрытый комплекс, в котором две цепи ДНК остаются спаренными по всей длине. На следующей стадии фермент катализирует открытие немногим более одного витка спирали ДНК, при этом образуется открытый комплекс, в котором матрица доступна для инициации синтеза цепи РНК. Полимераза, содержащая связанную с ней ситма-субъединицу, однако, ведет себя так, как будто она прикреплена к области промотора: она не способна осуществлять элонгацию цепи РНК и часто опять превращается в закрытый комплекс. Как показано на рисунке, для превращения фермента в полимеразу, активно осуществляющую элонгацию, необходимо, чтобы от него отделились факторы инициации (в случае Е. coli-сигма-фактора) и присоединились другие белки, выполняющие роль факторов элонгации (например, белок nusA у Е. coli). (С изменениями по D.C. Staney and D. M. Crothers, J. Мої. Biol. 193: 267-278, 1987.)
У всех организмов различные гены транскрибируются с разной скоростью. На некоторых промоторах новая цепь РНК инициируется каждые 1-2 секунды, тогда как на других для начала синтеза РНК требуется час. Обычно уровень транскрипции каждого гена определяется белками-регуляторами генетической активности, влияющими на инициацию транскрипции (гл. 10). В целом, действие этих белков основано на ускорении или замедлении одной или более стадий, указанных на рис. 9-65.
9-25
9.4.2. Синтез РНК у эукариот осуществляют три различные РНК-полимеразы [43]
Хотя транскрипция у эукариот и такого прокариота, как Е.coli, в чем-то схожа, в эукариотических клетках этот механизм гораздо сложнее. И у дрожжей, и у человека имеется три типа РНК-полимераз, каждый из которых ответствен за транскрипцию разных генов. Структура этих ферментов, обозначенных как РНК-полимеразы I, II и III, близка, некоторые их субъединицы одинаковы, а некоторые отличаются (рис. 9-66). Каждый из них сложнее РНК-полимеразы E.coli и, как полагают, содержит 10 или более полипептидных цепей. Наиболее важное различие между бактериальными и эукариотическими РНК-полимеразами состоит в следующем. У бактерий этот фермент связывается непосредственно с промотором, а у эукариот РНК-полимеразы присоединяются к промоторам только в присутствии дополнительных белковых факторов. Отчасти по этой причине лишь в 1979 г. удалось разработать системы, позволяющие изучать механизм инициации транскрипции у эукариот in vitro. Изначально три эукариотические РНК-полимеразы были идентифицированы на основании различия в их чувствительности к а-аманитину, высокотоксичному веществу, выделенному из бледной поганки. Как оказалось, а-аманитин не действует на РНК-полимеразу I, РНК-полимераза II к нему очень чувствительна, а у РНК-полимеразы III этот признак выражен умеренно. Чувствительность синтеза РНК к а-аманитину до сих пор используется для определения того, какой именно полимеразой транскрибируется ген. Так было установлено, что только РНК-полимераза II транскрибирует гены, РНК которых затем транслируется в белки. Две другие полимеразы синтезируют лишь те РНК, которые имеют структурные или каталитические функции: РНК-полимераза 1 синтезирует большие рибосомные РНК, РНК-полимераза III - разнообразные короткие стабильные РНК, включая малую рибосомную 5S-PHK и транспортные РНК. Однако большая часть малых РНК, образующих мяРНК, синтезируются полимеразой II.
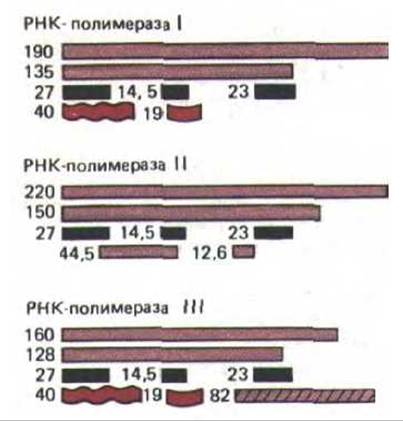
Рис. 9-66. Некоторые белковые субъединицы, входящие в состав трех эукариотических РНК-полимераз дрожжей. Показаны лишь наиболее хорошо изученные субъединицы. Родственные субъединицы окрашены одинаково, их молекулярная масса приведена в килодальтонах. Все три фермента имеют три субъединицы с одинаковой массой (выделены черным).
В клетках млекопитающих обычно содержится около 40 000 молекул РНК-полимеразы II, примерно такое же число РНК-полимеразы I и приблизительно 20000 молекул РНК-полимеразы III. Изучение культивируемых клеток показало, что концентрация РНК-полимераз регулируется пo-отдельности, в зависимости от скорости роста клетки.
9-26
9-27
9.4.3. Факторы транскрипции образуют стабильные комплексы на эукариотических промоторах [44]
Как отмечалось выше, РНК-полимеразы эукариот не узнают свои промоторы на очищенных молекулах ДНК. Чтобы это произошло, с ДНК должны связаться один или более сайт-специфических белков. Такие белки называют факторами транскрипции (TF). Они отличаются от сигма-факторов прокариот (а-факторы) тем, что могут связываться с ДНК независимо от РНК-полимеразы. Полимеразы I, II и III требуют присутствия разных факторов транскрипции, обозначенных TFI, TFII и TFIII соответственно. Латинская буква, которая обычно следует за римской цифрой в названии фактора транскрипции, указывает, каким по счету был выделен данный фактор. Например, TFIIIA - это первый охарактеризованный фактор транскрипции, который действует на ген, транскрибируемый полимеразой III (ген 5S-pPHK).
По-видимому, in vitro факторы транскрипции образуют довольно стабильные транскрипционные комплексы, которые избирательно притягивают молекулы РНК-полимеразы к своим промоторам. Различные факторы присоединяются к ДНК в разных местах относительно положения сайта начала транскрипции. Полимераза I и полимераза II образуют комплекс с тем фактором транскрипции, который связывается непосредственно перед сайтом начала транскрипции. Между тем основной фактор транскрипции для генов, считываемых полимеразой III, присоединяется к ДНК сразу за сайтом начала транскрипции, и таким образом, РНК-полимеразе III приходится осуществлять свою функцию, не смещая этот белок с ДНК (рис. 9-67). Полагают, что данный фактор (TFIIIC) накручивает ДНК на себя, образуя большую нуклеопротеиновую частицу.
О транскрипционной активности ДНК судят по её взаимодействию с РНК-полимеразой. При этом сравнивают молекулы ДНК, которые были предварительно проинкубированы с факторами транскрипции, и молекулы, не прошедшие такой обработки. Оказалось, что РНК-полимераза охотно использует для синтеза РНК молекулы ДНК, связанные с определенными факторами, тогда как во втором случае, хотя сами по себе молекулы ДНК идентичны первым, связывания РНК-полимеразы не происходит (рис. 9-68). Аналогичная ситуация характерна для молекул ДНК, инъецированных в ооцит Хепорт, следовательно, такой же механизм действует и in vivo.
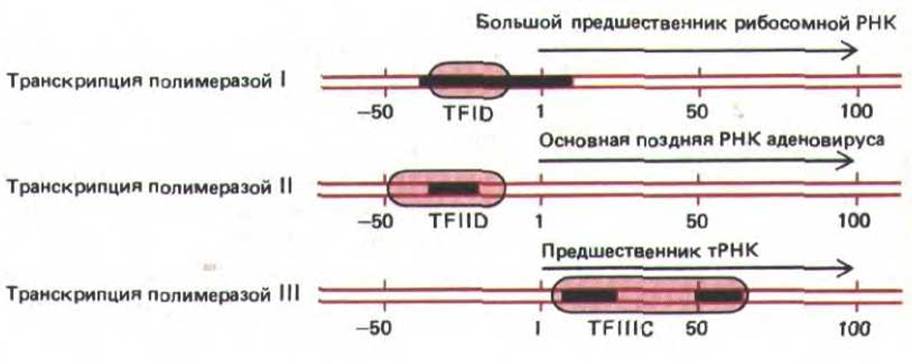
Рис. 9-67. Некоторые стабильные комплексы, образуемые факторами транскрипции с эукариотическими генами. Черным цветом обозначены определенные последовательности ДНК, которые, как полагают, необходимы для функционирования промоторов. У каждого гена цветом выделены области, которые «закрываются" присоединившимися факторами (на основании данных футпринтинга ДНК, см. рис. 4-69). Факторы транскрипции представляют собой молекулу белка, которая, вероятно, состоит из нескольких субъединиц, хотя доподлинно строение этих факторов неизвестно. Цифрами обозначено положение на молекуле ДНК (в нуклеотидных парах) относительно сайта начала транскрипции, принятого за +1.
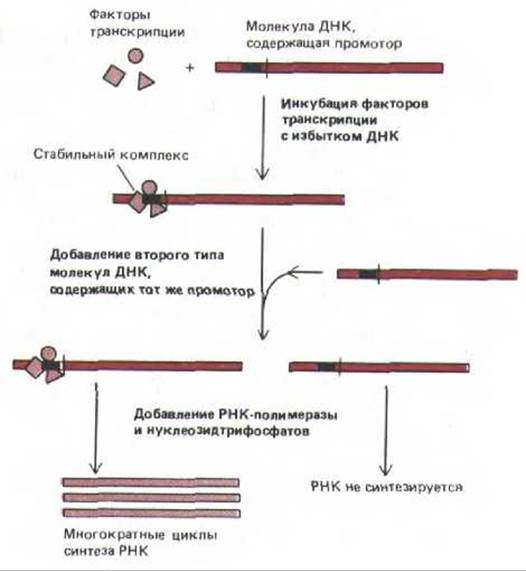
Рис. 9-68. Опыты in vitro, демонстрирующие важность образования стабильного транскрипционного комплекса на эукариотическом промоторе. Эксперименты проводили с факторами транскрипции и с промоторами, специфичными к каждой из трех эукариотических РНК-полимераз. Примеры клонированных генов, используемых в этих опытах, представлены на рис. 9-67.
Важным фактором транскрипции для многих промоторов РНК-полимеразы II служит TFIID. Он представляет собой большой белковый комплекс, который обычно называют ТАТА-фактор, так как он может связываться с консервативной АТ-богатой последовательностью, называемой ТАТА-бокс, которая расположена примерно за 25 нуклеотидов до сайта начала транскрипции. Механизм действия ТАТА-фактора, стимулирующего транскрипцию полимеразой II, представлен на рис. 9-69.
В связи с тем, что именно РНК-полимераза II отвечает за образование всех предшественников мРНК, и таким образом определяет, какие конкретно белки будут образовываться в клетке, в дальнейшем мы будем обсуждать синтез и судьбу транскриптов РНК, синтезируемых только этим ферментом.

Рис. 9-69. Минимальные условия для узнавания промотора молекулой эукариотической РНК-полимеразы II. Чтобы промотор был узнан полимеразой, необходимо, чтобы фактор связывания с ТАТА-боксом (TFIID) образовал стабильный транскрипционный комплекс. Консенсусная последовательность ТАТА-бокса следующая: Т82А97Т93А85 (А или Т)83; числа указывают на вероятность (в процентах) присутствия указанного нуклеотида. См. также рис. 10-27. (По J.2. Workman and R.G. Roeder, Cell 51: 613-622, 1987.)
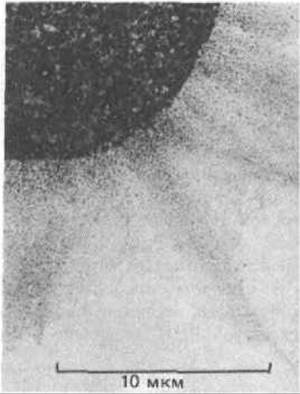
Рис. 9-70. Электронная микрофотография типичного препарата клеточного ядра. Видна невероятно сложная сеть из переплетенных нитей хроматина, высвободившегося из подвергнутого лизису ядра. Лишь на самом краю этой сети хроматин имеет достаточно низкую плотность, чтобы его можно было исследовать при большом увеличении. (С любезного разрешения Victoria Foe.)
9-28
9.4.4. РНК-полимераза ll транскрибирует некоторые последовательности ДНК гораздо чаще других [45]
Эксперименты, проводимые с очищенными препаратами подимераз и факторами транскрипции in vitro, безусловно важны для изучения условий протекания транскрипционных процессов, однако многие сведения о транскрипции ДНК могут быть получены только с помощью электронной микроскопии, которая позволяет наблюдать активные гены вместе с транскрибирующей их РНК-полимеразой.
Обычная электронная микроскопия тонких срезов выявляет лишь гранулированные глыбки хроматина, указывая на наличие развитой внутриядерной структуры (рис. 9-100), но дает очень мало информации о том, как именно транскрибируются гены. Гораздо более информативны в этом отношении снимки изолированного ядерного содержимого, непосредственно наносимого на электронномикроскопическую сеточку после разрушения ядер (рис. 9-70 и 9-71). На достаточном удалении от центра разрушенного ядра концентрация хроматина относительно низка, что позволяет рассмотреть индивидуальные хроматиновые фибриллы, имеющие характерный вид «бус на нитке».
Транскрипционно активные молекулы РНК-полимеразы выглядят как большие глобулярные частицы, за каждой из которых тянется длинная молекула новосантезированной РНК. При этом молекулы РНК-полимеразы II, как правило, представлены одиночными частицами. Это свидетельствует о том, что большинство генов транскрибируется в предшественники мРНК относительно редко, так что одна РНК-полимераза успевает завершить транскрипцию до того момента, когда ее начнет следующая. Впрочем, в некоторых случаях можно увидеть множество РНК-полимеразных частиц (и РНК-транскриптов), собранных в одном месте. Подобные кластеры типичны лишь для относительно небольшого числа генов, транскрибирующихся с высокой частотой (рис. 9-72). Длина молекул РНК, прикрепленных к такому кластеру, увеличивается в направлении движения молекул полимеразы, что придает всей структуре характерный вид. Каждый такой участок несет специфические сигналы начала и окончания транскрипции для РНК-полимеразы II и представляет собой транскрипционную единицу (рис. 9-73).
Биохимические исследования подтвердили результаты, полученные с помощью электронной микроскопии и позволили сформулировать три основных вывода.
1. Эукариотические РНК-полимеразы, как и прокариотические ферменты, начинают и оканчивают транскрипцию лишь в определенных точках хромосомы.
2. Средняя длина новообразованной молекулы РНК, синтезированной РНК-полимеразой на транскрипционной единице, составляет около 8000 нуклеотидов, однако» не редкость и молекулы РНК, насчитывающие от 10 до 20000 нуклеотидов. Это намного больше, чем 1200 нуклеотидов, т. е, больше той величины, которая достаточна для синтеза средней белковой молекулы, содержащей 400 аминокислотных остатков; такая большая длина молекул РНК отражает особенности строения эукариотических генов, которые мы подробно обсудим в дальнейшем.
3. Хотя большая скорость удлинения цепи (около 30 нуклеотидов в секунду) характерна для всех РНК, различные точки начала транскрипции для РНК-полимеразы II обладают очень разной эффективностью, и, таким образом, одни гены транскрибируются гораздо быстрее других. Данные электронномикроскопического исследования хорошо согласуются с результатами биохимического анализа, которые свидетельствуют.
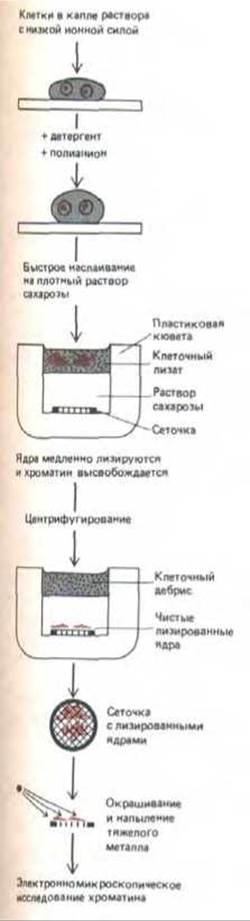
Рис. 9-71. Метод электронномикроскопического исследования хроматина, основанный на мягком лизисе и последующей очистке лизированного ядра от клеточных обломков.
о том, что хотя в клетке накапливается множество молекул мРНК различных типов, большая их часть представлена относительно малым числом копий (табл. 9-2).
9.4.5. Предшественники информационной РНК ковалентне модифицированы с обоих концов [46]
Транскрипты, синтезированные в ядре РНК-полимеразой II, называют гетерогенной ядерной РНК (мРНК), поскольку важнейшим признаком, отличающим эти молекулы от других ядерных РНК, служит гетерогенность их размеров. Многие гетерогенные ядерные транскрипты впоследствии покинут ядро, превратившись в молекулы информационной или матричной РНК (мРНК). Однако прежде чем выйти из ядра, молекулы мРНК претерпевают серию ковалентных модификаций, наделяющих эти молекулы свойствами, которые отличают их от транскриптов, синтезированных всеми другими РНК-полимеразами. Эти модификации окажутся им необходимыми позже, при функционировании в качестве мРНК в цитоплазме.
5'-конец молекулы РНК (конец, синтезируемый при транскрипции первым) прежде всего копируется, т. е. достраивается с образованием особой структуры, ответственной за последующее связывание молекулы мРНК с рибосомой. Кэпирование (добавление метилированного G-нуклеотида) происходит почти сразу после синтеза первых 30 нуклеотидов РНК и осуществляется путем соединения трифосфатной группы молекулы GTP с дифосфатом на 5'-конце первичного транскрипта (рис. 9-74). Помимо важной роли, которую играет 5'-кэп в инициации белкового синтеза, его функция состоит в том, чтобы защищать транскрипт РНК от деградации.
3'-конец большинства транскриптов, синтезируемых РНК-полимеразой II, образуется не в результате терминации транскрипции (соответствующий сайт расположен дальше), а в результате вторичной модификации, при которой растущий транскрипт расщепляется в определенном месте и к 3'-концу в точке разреза специальная полимераза добавляет polyA-последователыюсть. Сигналом к разрезанию служит появление на цепи РНК участка AAUAAA (за 10-30 нуклеотидов до сайта расщепления) и еще одной, хуже изученной последовательности. Сделав разрез, фермент роlуА-полимераза присоединяет от 100 до 200 остатков адениловой кислоты (так называемый роlуА-«хвост") к 3'-концу цепи РНК, чем завершает образование первичного транскрипта РНК, А тем временем РНК-полимераза продолжает транскрипцию и делает это до тех пор, пока не встретит на своем пути сайт терминации. Но дополнительные фрагменты транскрипта РНК лишены кэпа и, вероятно, поэтому быстро распадаются (рис. 9-75).
Функция роlуА-«хвоста" до сих пор не очень ясна. Согласно одной из гипотез, эта последовательность участвует в транспорте зрелой мРНК из ядра. Имеются также данные, свидетельствующие о том, что polyA-конец замедляет деградацию в цитоплазме некоторых молекул мРНК, что способствует их стабилизации.
Хотя транскрипты, синтезированные РНК-полимеразой II, составляют более половины РНК, образующейся в клетке, мы уже убедились в том, что большая часть РНК этих транскриптов нестабильна и, следовательно, существует в клетке недолго. В итоге, мРНК, содержащаяся в ядре, и образовавшаяся из нее цитоплазматическая мРНК составляют лишь малую часть всей РНК клетки (табл. 9-3). Несмотря на то, что молекулы мРНК относительно редки, выделить их довольно легко благодаря присутствию на 3'-конце длинной роlуА-последовательности. Если пропустить всю РНК клетки через колонку, заполненную электронномикроскопических данных можно определить направление синтеза цепи РНК и идентифицировать сайты начала и конца этой единицы. polyT-связанным матриксом, молекулы с роlуА-«хвостом" будут избирательно задерживаться на такой колонке вследствие комплементарного спаривания Т и А. Этот метод широко используется для отделения гяРНК и мРНК от молекул рибосомной и транспортной РНК, составляющих основную массу РНК клетки.
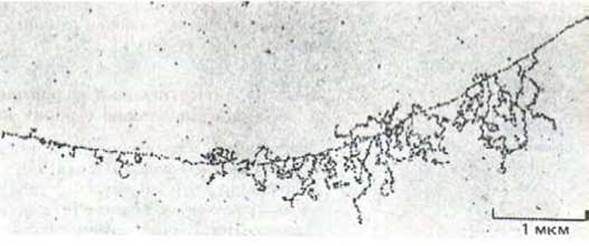
Рис. 9-72. Необычная область хроматина, в которой один из генов транскрибируется с высокой частотой; хорошо видно большое число молекул РНК-полимеразы II вместе с растущими цепями РНК-транскриптов. Транскрипция направлена слева направо (см. рис. 9-73). По V. Е. Foe, Z. Е. Wilkinson and CD. Zaird, Cell 9: 131-146. © 1976 Cell Press.)

Рис. 9-73. Схематическое изображение идеальной транскрипционной единицы, показывающее, как на основе
9-30
9.4.6. Для кэпирования и добавления polyA требуется РНК-полимераза II [47]
5'-Кэпы и 3'-роlуА-последовательности имеются только в тех молекулах, которые транскрибированы РНК-полимеразой II. Методы генетической инженерии сегодня позволяют отделить ген, транскрибируемый в норме РНК-полимеразой II, от своего промотора и поставить под промотор, узнаваемый РНК-полимеразами I или III. Оказалось, что если такой модифицированный ген вновь ввести в клетку, то он будет транскрибироваться одной из этих полимераз, но при этом вновь синтезированные молекулы РНК не будут ни кэпированы, ни полиаденилированы. Следовательно, и кэпирование, и полиаденилирование осуществляются с участием ферментов, которые избирательно связываются с РНК- полимеразой II и активны лишь в комплексе с ней. Такие ферменты можно рассматривать как факторы элонгации РНК-полимер азы II. Вероятно, именно необходимость кэпировать и полиаденилировать предшественники мРНК можно объяснить, почему у эукариот эти молекулы синтезируются специальной РНК-полимеразой.
Таблица 9-2. Популяция молекул мРНК в типичной клетке млекопитающих
|
Класс |
Общее число копий каждой индивидуальной последовательности мРНК на клетку |
Число индивидуальных последовательностей мРНК в каждом классе |
Общее число молекул мРНК в каждом классе |
|
Богатый |
12000 |
X 4 |
= 48 000 |
|
Средний |
300 |
X 500 |
= 150000 |
|
Бедный |
15 |
X 1 1 000 |
= 165000 |
Подобное деление клеточной мРНК на три дискретных класса в известной степени искусственное: в ряде случаев наблюдается более плавное распределение различных молекул мРНК в соответствии с их содержанием в клетке. В сумме в каждой нормальной клетке насчитывается от 10000 до 20000 разных видов мРНК, большинство из которых представлено малым числом копий (от 5 до 15 молекул на одну клетку). Обычно общая цитоплазм этическая РНК представлена в основном рРНК, а на долю мРНК остается всего лишь от 3 до 5%, так что на одну молекулу мРНК приходится примерно 10 рибосом. Клетка данного типа содержит в сумме около 360 000 молекул цитоплазматической мРНК.
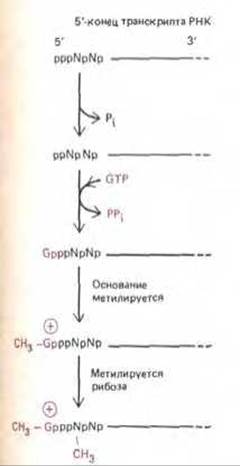
Рис. 9-74. Кэпирование 5'-концов молекул РНК, синтезированных РНК-полимеразой II. Присоединенный кэп содержит новую связь 5' → 5' между положительно заряженным остатком 7-метил-О- и исходным 5'-коицом транскрипта РНК (см. рис. 5-24). Полагают, что по крайней мере некоторые ферменты, участвующие в этом процессе, связаны с полимеразой II, так как транскрипты, образуемые полимеразами I и II не кэпируются. Буквой N обозначается любой из четырех рибонуклеотидов, хотя цепь РНК обычно начинается с пурина (А или G). (По A, J. Shatkin, Bioessays 7: 275-277, 1987.)
О том, что для реакции разрезания и полиаденилирования, по-видимому, необходим фактор элонгации, связанный с РНК-полимеразой, свидетельствовали следующие факты. В нескольких работах было показано, что каждая транскрибирующая молекула РНК-полимеразы II способна образовать только один полиаденилированный 3'-конец, т. е. все сигналы AAUAAA, встречаемые полимеразой после первого такого сайта, игнорируются. Это дает основание для гипотезы, что полимераза содержит фактор, который после завершения реакций разрезания и полиаденилирования, теряется (рис. 9-75).
9.4.7. Большая часть РНК, синтезированной РНК-полимеразой II, в ядре быстро распадается [48]
Первые данные о нестабильности в ядре транскриптов, синтезированных РНК-полимеразой II, были получены в опытах на культуре клеток. В среду для культивирования на короткое время вводили [3Н]-уридин. При этом радиоактивная метка включается в молекулы гяРНК, за судьбой которых затем можно следить в течение более длительного времени. Эти эксперименты позволили сформулировать два важных вывода.
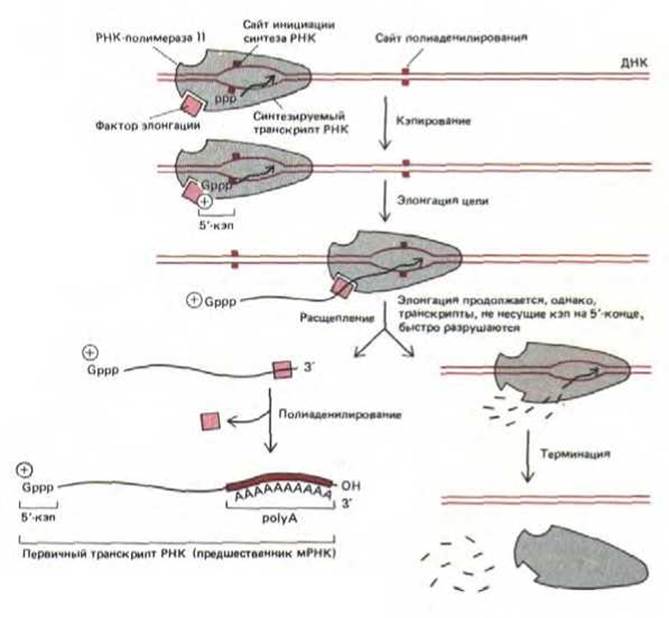
Рис. 9-75. Синтез молекулы гяРНК (предшественника мРНК) с помощью РНК-полимеразы II. Полимераза на рисунке только начала синтез цепи РНК (стадия 5, рис. 9-65). Узнавание дополнительного сигнала полиаденилирования на растущем транскрипте РНК приводит к разрыву и полиаденилированию цепи. У дрожжей полимераза сразу же после этого заканчивает синтез РНК, однако у высших эукариот транскрипция часто продолжается. Представляется вероятным, что свойства полимеразы меняются, как только происходит расщепление одной цепи РНК; таким образом, полимераза не может участвовать в полиаденилировании транскриптов, считываемых после этого. Весьма возможно, что в таком виде полимераза лучше распознает последовательности, определяющие терминацию и высвобождение полимеразы. Самое простое объяснение, представленное на этом рисунке, заключается в том, что после разрезания транскрипта от полимеразы отделяется фактор (или факторы) элонгации.
Таблица 9-3. Содержание различных видов РНК в типичной клетке млекопитающих.

Приведенные данные получены на основе анализа культивируемых фибробластов мыши (L-клетки). Каждая такая клетка содержит 26 пг РНК (5 х 10110 рибонуклеотидов), причем 14% этого количества находится в ядре. (Таким образом, ядро клетки содержит вдвое больше ДНК, чем РНК.) Это приблизительно в 20 раз превышает среднюю скорость синтеза ДНК в S-фазе. Следует подчеркнуть, что хотя гяРНК составляет большую часть всей синтезируемой в клетке РНК, лишь очень небольшое ее количество избегает деградации в ходе ядерного процессинга РНК. Поэтому образующаяся из гяРНК мРНК представляет собой минорную фракцию клеточной РНК (по Brandhorst В. P., McConkey Е. Н. J. Мої. Biol. 85; 451563, 1974)
1. Длина вновь синтезированных молекул гяРНК быстро уменьшается, достигая размеров цитоплазматической РНК примерно за 30 минут. Первичные транскрипты РНК содержат в среднем около 6000 нуклеотидов, тогда как длина молекул мРНК составляет приблизительно 1500 нуклеотидов.
2. Примерно через 30 минут радиоактивно меченные молекулы РНК начинают покидать ядро в виде молекул мРНК. Однако всего лишь около 5% всей меченой гяРНК достигает цитоплазмы клетки. Оставшиеся молекулы примерно в течение часа распадаются в ядре на маленькие фрагменты. Когда в начале 70-х гг. было обнаружено, что и мРНК, и гяРНК на 3'-конце содержат polyA, естественно было предположить, что мРНК образуется путем значительной деградации гяРНК на 5'-концах, другими словами, что молекулы гяРНК состоят из очень длинной «5'-лидерной последовательности», расположенной перед кодирующей частью. Однако от этой гипотезы пришлось отказаться, когда на 5'-конце были обнаружены кэпы, которые, как выяснилось, при превращении молекул гяРНК в мРНК по большей части сохраняются. По-видимому, уже тогда можно было догадаться о том, что удаляется середина молекулы РНК, а ее 3'- и 5'-концы остаются интактными. Но в то время такое предположение сочли бы абсурдным. Более того, исследователи терялись в догадках, почему клетка должна избавляться от большей части вновь синтезированной РНК. Эта загадка была разгадана лишь тогда, когда стало возможным сравнивать нуклеотидную последовательность отдельной молекулы мРНК с последовательностью кодирующей ее геномной ДНК.
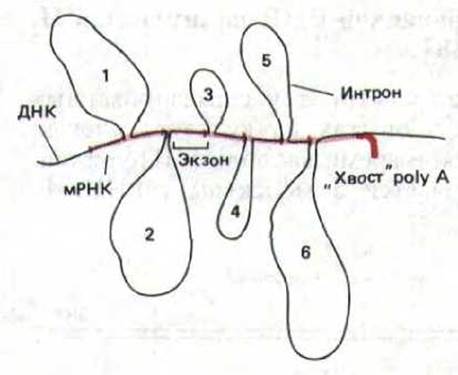
Рис. 9-76. Первые данные, свидетельствующие о существовании интронов в генах эукариот, получены при использовании метода «R-петель». Комплекс, содержащий спаренные основания молекул мРНК и ДНК, визуализировали с помощью электронного микроскопа. Молекулы мРНК, содержащиеся в очень больших количествах, такие, как мРНК р-глобина или овальбумина, легко выделить из синтезирующих их специализированных клеток. При гибридизации таких препаратов одноцепочечной мРНК с клонированной двухцепочечной ДНК, которая содержит ген, кодирующий эту мРНК, РНК может заместить ДНК на участке гомологии с образованием спирали РНК-ДНК. Области, в которых спаривания не произошло, хорошо различимы: они выглядят как большие петли. Каждая из этих петель, пронумерованных от 1 до 6, представляет собой
интрон, входящий в состав последовательности гена.
9.4.8. В ходе процессинга РНК из середины молекулы удаляются длинные последовательности нуклеотидов [48]
Открытие в 1977 г. прерывистых генов было совершенно неожиданным. Предшествовавшее этому событию многолетнее изучение бактерий привело исследователей к выводу, что гены состоят из непрерывных последовательностей, необходимых для кодирования аминокислотных остатков белка. Не было никаких причин полагать, что гены эукариот могут быть организованы как-то иначе. Первое указание на то, что в отличие от генов бактерий гены эукариот не являются непрерывными, получено при изучении РНК, синтезируемых аденовирусом человека (крупный ДНК-содержащий вирус). Оказалось, что в той области вирусной ДНК, на которой синтезируются эти мРНК, присутствуют последовательности, никогда не встречающиеся в зрелой РНК. Предположение о том, что это явление присуще только вирусам, было быстро отвергнуто после того, как аналогичную прерывистость обнаружили в генах овальбумина и ß-глобина позвоночных. Как уже упоминалось выше, последовательности, присутствующие в ДНК, но не входящие в состав мРНК, были названы нитронами, а последовательности, присутствующие в зрелой мРНК, - экзонами (рис. 9-76 и 9-77).
Теперь известно, что первичный транскрипт РНК - это точная копия гена, содержащая как экзоны, так и интроны. Последовательности нитронов вырезаются из середины транскрипта РНК, в результате чего образуется молекула мРНК, непосредственно кодирующая белок (рис. 3-13). Поскольку кодирующие последовательности с обеих сторон интрона после его удаления соединяются друг с другом, эту реакцию назвали сплайсингом РНК (to splice-сращивать). Сплайсинг РНК протекает в клеточном ядре вдали от рибосом и РНК переносится в цитоплазму только после завершения этого процесса.
В настоящее время известно, что гены млекопитающих содержат гораздо больше интронных последовательностей, чем экзонов (см. табл. 9-1). Вот почему очень длинные молекулы гяРНК (более 50000 нуклеотидов) после сплайсинга превращаются в более короткие молекулы цитоплазматической мРНК (от 500 до 3000 нуклеотидов).
Перед тем как обсудить закономерности распределения нитронов внутри эукариотических генов, а также их значение для клеток эукариот, необходимо объяснить, как последовательности нитронов узнаются и удаляются при сплайсинге.
9-31
9.4.9. К транскриптам гяРНК сразу же присоединяются белки и мяРНП [49]
У эукариот в отличие от бактерий, вновь синтезированная РНК, по-видимому, сразу же конденсируется с образованием множества рядом расположенных частиц, содержащих белок. В состав этих частиц входит РНК длиной приблизительно 5000 нуклеотидов, нить которой намотана на белковый остов. Гетерогенное ядерные рибонуклеопротеиновые частицы (гяРНП-частицы) можно выделить путем обработки ядер рибонуклеазой. Концентрация фермента должна быть подобрана таким образом, чтобы разрушить РНК, связывающую эти частицы. Константа седиментации гяРНП-частиц около 30S, а их диаметр примерно в два раза превышает диаметр нуклеосом (20 нм). Соответственно и белковая сердцевина этих частиц больше и сложнее по составу, в нее входит по крайней мере 8 различных белков массой от 34000 до 120000 дальтон. Все они принадлежат к классу наиболее часто встречающихся белков клеточного ядра. Охарактеризованные к настоящему времени белки этой частицы, как оказалось, содержат одну или более копий короткой последовательности аминокислот, которая часто встречается в составе многих белков, связывающихся с РНК (рис. 9-78).
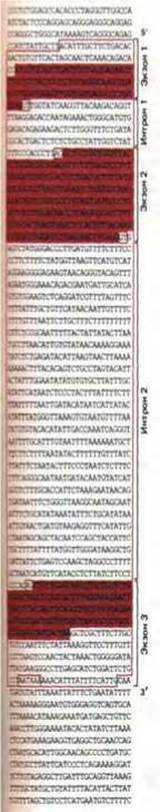
Рис. 9-77. Транскрибируемая часть гена р-глобина человека. Приведена последовательность той цепи ДНК, которая соответствует последовательности мРНК. Первичный транскрипт РНК обведен цветной линией, а нуклеотиды, входящие в состав трех кодирующих областей (экзонов), выделены цветом. Следует отметить, что экзон 1 содержит 5'-лидерную последовательность, а экзон У включает в себя 3'-трейлерную (дополнительную) последовательность. Хотя эти последовательности и входят в состав мРНК, они не кодируют аминокислоты. Высококонсервативные нуклеотиды GT и AG на концах каждого интрона обведены (см. рис. 9-79); обведены также сигналы разрезания и полиаденилирования вблизи 3'-конца гена (ААТААА, см. рис. 9-75).
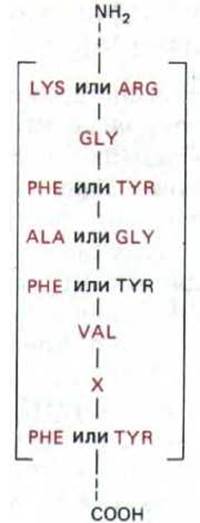
Рис. 9-78. Аминокислотная последовательность, присутствующая во многих эукариотических РНК-связывающих белках. Эта последовательность обнаруживается у столь различных организмов, как дрожжи, дрозофила и человек. Она входит в состав белков гяРНП-частиц; белков, связывающихся с роlуА-«хвостом» гяРНК; белков, образующих мяРНП; присутствует в нуклеолине-белке, который в избытке имеется в ядре. Выявление такой последовательности в белке, функция которого неизвестна, дает основание предполагать, что этот белок связывается с РНК.
Применение стандартных методик распластывания, благодаря которым транскрибирующиеся гены можно наблюдать под электронным микроскопом, к сожалению, часто приводит к разрушению частиц гяРНП (см. рис. 9-70). Тем не менее на этих препаратах удается обнаруживать необычного вида частицы. Их расположение достаточно убедительно доказывает участие в сплайсинге РНК. Стабильные частицы очень быстро образуются в местах соединения последовательностей нитронов и экзонов и по мере элонгации транскрипта РНК они сливаются в пары с образованием больших агрегатов. Предполагают, что эти агрегаты представляют собой сплайсосомы, катализирующие сплайсинг РНК (рис. 9-79).
Биохимический анализ выявил в клеточном ядре еще один класс частиц, состоящих из белка и маленьких молекул РНК (250 нуклеотидов или меньше), которые произвольно были обозначены U1, U2, ..., U12-PHK. Эти комплексы, названные малыми ядерными рибонуклеопротеинами (мяРНП), напоминают рибосомы в том отношении, что каждая из этих частиц содержит набор белков, связанных со стабильной молекулой РНК. По размеру, однако, эти частицы гораздо меньше рибосом (около 250000 дальтон по сравнению с 4,5 млн. дальтон рибосомы), кроме того, в этих частицах отношение белок/РНК выше, чем у рибосом. Некоторые белки входят в состав различных типов мяРНП, другие присуши только одному типу частиц. Впервые это удалось показать в опытах с сывороткой крови, полученной от больных системной красной волчанкой. У таких больных в крови имеются антитела к одному или более белку, входящему в состав мяРНП. Например, оказалось, что одно и то же антитело связывает U1-, U2--, U5- и U4/б-мяРНП. По-видимому, это объясняется тем, что в их состав входит один и тот же белок. Полагают, что отдельные мяРНП на основе комплементарности узнают специфические последовательности нуклеиновой кислоты. Некоторые частицы, вероятно, участвуют в сплайсинге РНК; какие-то мяРНП вовлечены в реакции расщепления, в ходе которых образуется 3'-конец вновь синтезированных РНК; функция других мяРНП неизвестна. Доказательства участия мяРНП в сплайсинге получены в опытах по изучению процессинга РНК in vitro.
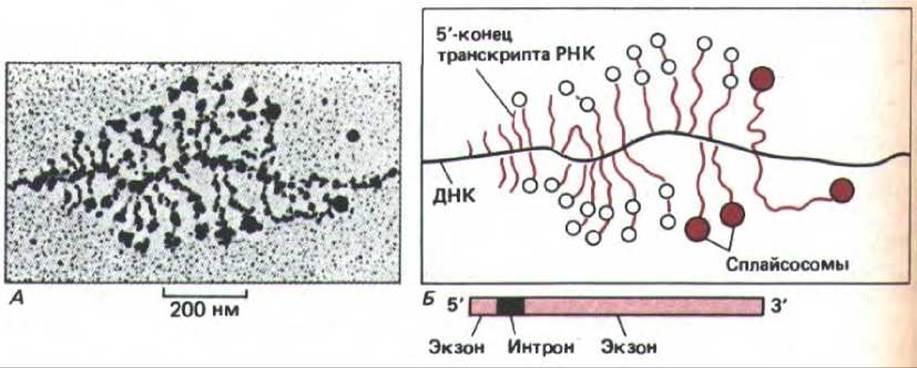
Рис. 9-79. Электронная микрофотография растянутого хроматина. Видны большие рибонуклеопротеиновые частицы, собирающиеся на 5'- и 3'-сайтах сплайсинга и образующие сплайсосомы. На фотографии (А) представлен ген, кодирующий белок хориона дрозофилы; положение сайтов сплайсинга на его первичных транскриптцх известно. Б. Большинство транскриптов РНК вблизи своих 5'-концов содержит одну или две большие РНП-частицы. Если на транскрипте присутствует две частицы (пустые кружки на схеме Б), то они, как правило, имеют диаметр 25 нм и расположены вблизи или в самих 5'- и 3'-сайтах сплайсинга отдельных небольших последовательностей нитронов (длиной 228 нуклеотидов) в области У-концов транскриптов. Более длинные, более зрелые транскрипты двух генов часто несут единственную частицу большего размера (цветные кружки на схеме Б) в области интрона. Вероятно, она возникает в результате стабильной ассоциации двух меньших частиц и представляет собой объединенную сплайсосому. Поскольку в ряде случаев сплайсинг происходит тогда, когда 3'-конец РНК еще транскрибируется, можно сделать вывод, что для сплайсинга не нужна polyA-последовательность на 3'-конце молекулы гяРНК. (С изменениями по I. N. Osheim, О. Z. Miller and A.Z. Beyer, Cell 43: 143-151, 1985.)

Рис. 9-80. Консенсусные последовательности 5'- и 3'-сайтов сплайсинга РНК. Приведенная последовательность дана для цепи РНК. Обязательные динуклеотиды GU и AG на каждом конце интрона выделены цветом (см. также рис. 9-77).
9.4.10. Последовательности нитронов удаляются в виде лассо-подобных РНК-структур [50]
Длина нитронов варьирует от 80 до 1000 и более нуклеотидов. Интроны принципиально отличаются от экзонов тем, что для них, по- видимому, не так важна строго определенная последовательность нуклеотидов. В ходе эволюции, вероятно, именно в них накапливаются мутации; часто удается изменить большую часть последовательности интрона, не затрагивая активности гена. На основании этих данных было высказано предположение, что последовательности нитронов функционально неактивны и служат главным образом генетическими «связками». Единственными высококонсервативными областями в интронах являются последовательности, необходимые для их удаления. На каждом конце

Рис. 9-81. Метод получения РНК, пригодной для анализа сплайсинга РНК in vitro. Прежде всего, необходимо иметь большое количество соответствующей последовательности ДНК (сконструировать ее методом генетической инженерии и клонирования); кроме того, нужны относительно простые РНК-полимеразы из бактериофагов Т7 или SP6, с высокой активностью транскрибирующие РНК in vitro. Если соединить фрагмент эукариотической ДНК с промотором бактериофага, то РНК-полимераза может синтезировать in vitro большое количество РНК, кодируемой фрагментами эукариотической ДНК. 5'-кэп, входящий в состав гяРНК и необходимый для инициации транскрипции, можно присоединить к таким РНК, используя искусственно синтезированный кэпированный нуклеотид (не показано).
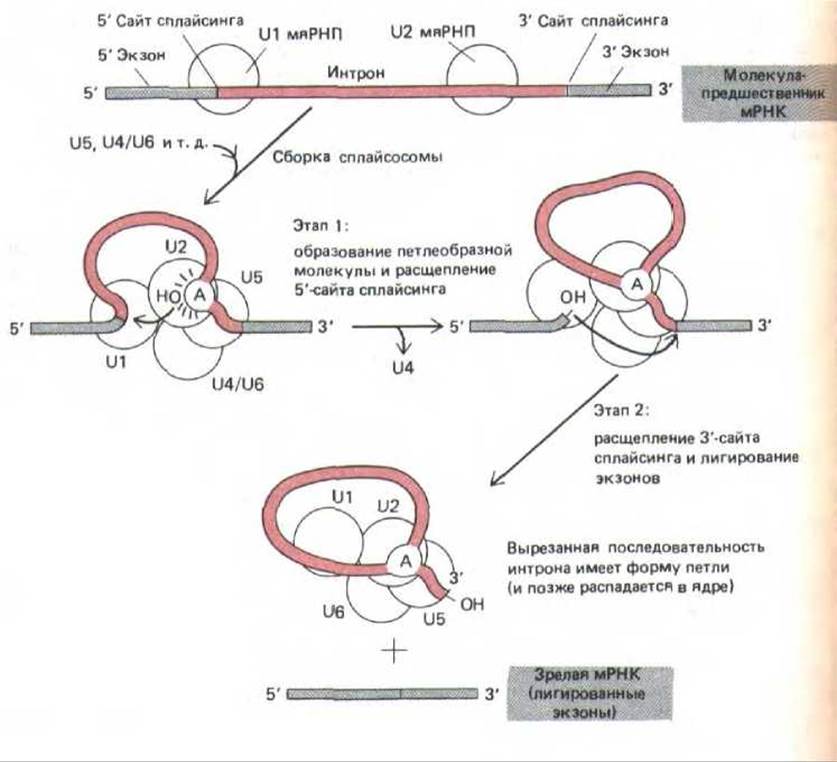
Рис. 9-82. Катализ сплайсинга РНК сплайсосомой, образовавшейся при соединении U1-, U2-, U5- и U4/6-мяРНП (обозначены кружочками), и других компонентов (не указаны). После сборки сплайсосо-мы реакция проходит в две стадии: на стадии 1 определенный А-нуклеотид, расположенный в последовательности интрона вблизи 3'-сайта сплайсинга, атакует 5'-сайт сплайсинга, который в результате разрезается.
Разрезанный 5'-конец последовательности интрона ковалентне связывается с этим А-нуклеотидом, образуя разветвленную структуру, представленную на рис. 9-83. На стадии 2 3'-ОН-конец первого экзона, образовавшийся на первой стадии, соединяется с началом второго экзона, разрезая при этом молекулу РНК по 3'-сайту сплайсинга. Таким образом, две последовательности экзонов соединяются вместе, а последовательность интрона высвобождается в виде лассо-подобноЙ структуры. Полный комплекс, образующий сплайсосому седиментирует при 60S; следовательно, по размерам он приближается к рибосоме. Процессинг РНК происходит в ядре, в результате этих событий из первичных РНК-транскриптов (предшественники мРНК) образуются молекулы мРНК.
интрона имеются консенсусные области, которые во всех нитронах почти одинаковы. Изменение этих последовательностей влияет на процесс сплайсинга, в результате которого из первичного транскрипта РНК удаляются последовательности нитронов. На рис. 9-80 показаны такие консервативные последовательности для 5’-сайта сплайсинга (донорный сайт) и 3’-сайта сплайсинга (акцепторный сайт). Реакция разрыва и воссоединения РНК должна происходить абсолютно точно, так как ошибка даже в один нуклеотид сместит рамку считывания в образовавшейся молекуле РНК и сделает бессмысленной закодированную в ней информацию.
Механизм удаления нитронов из первичных транскриптов анализировали в опытах in vitro. Для этого соответствующим образом сконструированную ДНК инкубировали с высокоочищенной РНК-полимеразой для получения индивидуальных РНК, содержащих единственный нитрон (рис. 9-81). Когда такие молекулы РНК добавляли к клеточному экстракту, происходил сплайсинг. Реакция носила одноступенчатый характер, для нее были необходимы продолжительная инкубация с АТР, наличие определенных белков в экстракте и мяРНП U1, U2, U5 и U4/6. Эти составляющие соединяются с образованием многокомпонентного рибонуклеопротеинового комплекса или сплайсосомы. Анализ РНК, которые являются промежуточными продуктами в этой реакции, а также мяРНП, необходимых для их образования, привел к следующему выводу: вырезание интрона сопровождается образованием лассо-подобной структуры (рис. 9-82 и 9-83).
В последнее время уточнена функция ряда мяРНП. Например, мяРНП U1 связывается с 5'-сайтом сплайсинга благодаря нуклеотидной последовательности в РНК U1, которая комплементарна консенсусной последовательности сигнала сплайсинга, состоящей из девяти нуклеотадов (см. рис. 9-80). Поскольку известно, что молекула РНК обладает ферментативной активностью (см. разд. 3.2.11), катализировать разрыв и образование ковалентных связей, необходимых для сплайсинга РНК, может либо она сама, либо белковый компонент сплайсосомы.

Рис. 9-83. Строение разветвленной цепи РНК, образующейся при сплайсинге РНК. А-нуклеотид, выделенный цветом, - это тот же самый нуклеотид, который фигурировал на рис. 9-82; здесь показано ответвление, образующееся на первой стадии реакции сплайсирования. На этой стадии 5'-конец последовательности интрона разрезается и его фосфатная группа ковалентно связывается с 2'-ОН-рибозной группой А-нуклеотида, расположенного на расстоянии 30 нуклеотидов от 3'-конца последовательности интрона. Разветвленная цепь остается в вырезаемой последовательности интрона и обусловливает его лассо-образную форму (см. рис. 9-82).
9-32
9.4.11. Из каждого транскрипта РНК обычно удаляется несколько интронных последовательностей [51]
В связи с тем, что сплайсосома главным образом узнает консенсусную последовательность на границах интрона, существует опасность, что 5'-донорный сайт на конце любого интрона соединится с 3'-сайтом сплайсинга не своего, а другого интрона. Когда 5'- и 3'-половинки двух разных нитронов в эксперименте соединяют вместе, получающиеся в результате последовательности гибридного интрона узнаются ферментом РНК-сплайсинга и удаляются.
Принимая во внимание эти данные может показаться странным, что гены позвоночных содержат иногда до 50 нитронов (см. табл. 9-1). Ведь если случайно по ошибке соединятся любые два 5'- и 3'-сайта сплайсинга, это приведет к потере неких функционально активных последовательностей мРНК, что может иметь катастрофические последствия. Каким-то образом подобные ошибки не допускаются: механизм процессинга в норме гарантирует, что каждый 5'-сайт сплайсинга спарится именно с ближайшим к нему 3'-сайтом сплайсинга, расположенным в направлении от 5' к 3' на линейной последовательности РНК (рис. 9-84). Пока неизвестно, как осуществляется такое последовательное соединение. Вероятно, важную роль в этом играет сборка сплайсосом в ходе роста транскрипта РНК (см. рис. 9-79). Имеются также данные, свидетельствующие о том, что для правильного спаривания сайтов сплайсинга очень важна точная трехмерная конформация последовательностей интрона и экзона.
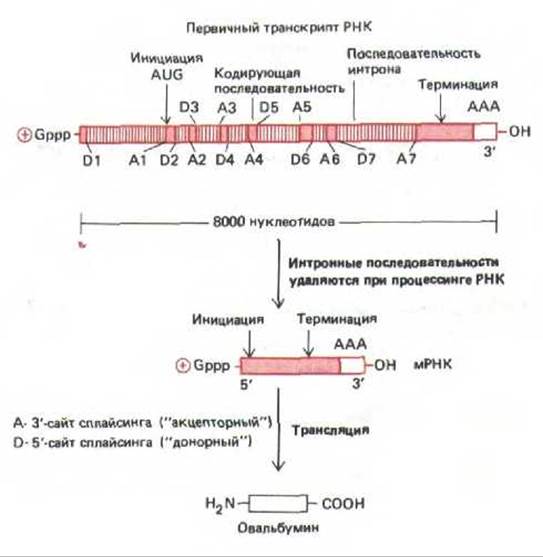
Рис. 9-84. Первичный транскрипт гена овальбумина курицы. Для получения активной молекулы мРНК из него удаляется семь нитронов. 5'-сайты сплайсинга (донорные сайты) обозначены буквой D, а 3'-сайты сплайсинга (акцепторные сайты) обозначены буквой А.
9-33
10-23
9.4.12. Один и тот же транскрипт РНК может сплайсироваться по-разному, при этом образуются мРНК, кодирующие разные белки [52]
Хотя большинство интронных последовательностей сами по себе, по-видимому, не обладают специфическими функциями, в ходе сплайсинга могут образоваться несколько различных мРНК. Следовательно, один и тот же транскрипт способен дать начало нескольким различным белкам, что сообщает клетке дополнительную генетическую пластичность. В гл. 10 будет показано, что в процессе дифференцировки клетки происходит изменение схемы сплайсинга многих транскриптов РНК, и таким образом по мере развития клетки, одни и те же кодирующие последовательности ДНК могут использоваться в разных целях (см. разд. 10.4.2).
Пластичность, обусловленная сплайсингом РНК, впервые была обнаружена у аденовирусов, у которых, собственно, и был открыт этот процесс. Геном аденовирусов детерминирует синтез очень длинных транскриптов РНК, кодирующих различные белки. В нормальной эукариотической клетке этого не происходит. Здесь каждая отдельная молекула мРНК кодирует лишь один белок, трансляция инициируется только вблизи 5'-кэпа и останавливается на первом же терминирующем кодоне. Однако у аденовируса существует определенный механизм сплайсинга РНК, который может использовать кодирующие последовательности в качестве нитронов и удалять их, при этом один и тот же 5'-кэп способен соединяться с любой из лежащих за ним кодирующих последовательностей, образуя различные мРНК в соотношениях, необходимых для жизнедеятельности вируса. Такой альтернативный сплайсинг РНК дает возможность использовать один и тот же 5'-кэп в качестве сигнала инициации синтеза различных белков (рис. 9-85). Этот способ широко используется вирусами, что позволяет небольшому числу различных транскриптов РНК кодировать значительное количество белков.
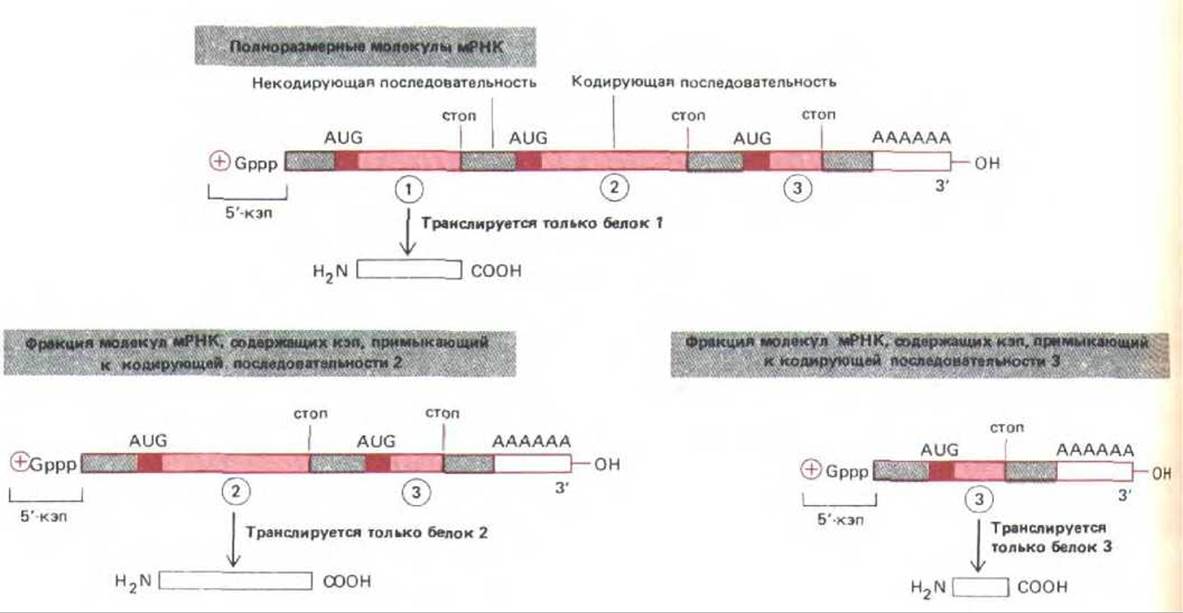
Рис. 9-85. У некоторых вирусов один и тот же первичный транскрипт сплайсируется по-разному, при этом образуется три (или более) молекулы мРНК, каждая из которых кодирует свой белок. Во всех случаях транслируется лишь кодирующая последовательность, примыкающая к 5'-кэпу.
9-34
9.4.13. Изменения мРНК при талассемии демонстрируют механизм возникновения новых белков при сплайсинге [53]
Благодаря методам генной инженерии исследователи получили возможность использовать для изучения клеточных механизмов мутации человека. Например, известно, что группа наследственных заболеваний крови, объединяемых под названием талассемии, характеризуется резким падением уровня гемоглобина. Секвенирование ДНК 50 больных талассемией показало, что в большинстве случаев снижение уровня гемоглобина было вызвано нарушением в сплайсинге РНК. Единичные замены нуклеотидов, обнаруженные в ДНК, либо инактивировали сайт сплайсинга, либо приводили к возникновению нового такого сайта. Удивительно, но анализ мРНК этих же больных показал, что потеря сайта сплайсинга не ведет к его отменению; оставшийся нормальным второй, участвующий в сплайсинге сайт, «ищет» поблизости подходящий участок и соединяется с ним. При этом может реализоваться несколько вариантов сплайсинга, т. е. мутантный ген способен детерминировать несколько измененных белков (рис. 9-86). Таким образом, сплайсинг представляет собой весьма пластичный процесс. Итак, мутация в эукариотической клетке может привести к тому, что с одного гена будет синтезироваться несколько разных новых белков, следовательно, клетка обладает возможностью весьма эффективно опробовать заложенные в ней генетические варианты. Благодаря этому сплайсинг РНК может играть решающую роль в эволюции высших эукариот. Сплайсинг у низших эукариот, например дрожжей, регулируется значительно более строго, что ограничивает вероятность появления новых мРНК в результате нарушения этого процесса. Следовательно, скорость дивергенции форм и функций у низших эукариот должна быть более медленной, чем у высших эукариот.
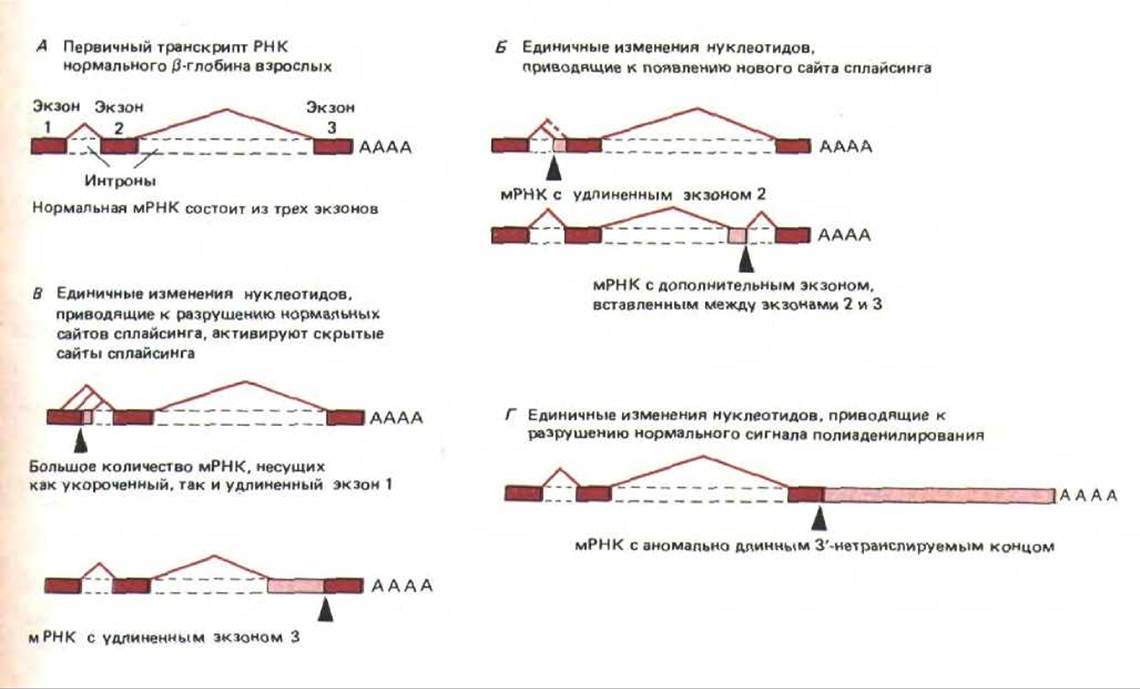
Рис. 9-86. Примеры аномального процессинга первичных транскриптов РНК р-глобина у больных талассемией. Мутантные сайты обозначены черными стрелками. Цветные прямоугольники соответствуют трем нормальным экзонам, изображенным на рис. 9-77. Цветные линии соединяют 5'- и 3'-сайты сплайсинга, участвующие в процессинге первичного транскрипта РНК. Незакрашенные участки представляют новые последовательности нуклеотидов, которые включаются в конечную молекулу мРНК в результате мутации. Следует отметить, что если при мутации нормальный сайт сплайсинга остается без партнера, то в качестве партнера используется один из аномальных «скрытых» сайтов сплайсинга, расположенных рядом. (По S. Н. Orkin, In: The Molecular Basis of Blood Diseases. [G, Stanato-gannopoulos et al., eds.] pp. 106-126, Philadelphia: Saunders, 1987.)
9.4.14. Сплайсинг РНК, катализируемый сплайсосомами, возможно, возник из самосплайсинга [54]
Открытие промежуточных структур, имеющих форму лассо, при сплайсинге ядерной РНК озадачило молекулярных биологов. Зачем клетке использовать такой причудливый способ сплайсинга, почему в ней не происходит простого соединения соответствующих 5'- и 3'-сайтов? По- видимому, ответ на этот вопрос можно найти, рассмотрев путь возникновения сплайсосом.
Согласно гипотезе, о которой шла речь в гл. 1, первые клетки в качестве основных катализаторов использовали молекулы РНК, а не белка, и генетическую информацию хранили в виде РНК, и не ДНК. Возможно, сплайсинг, катализируемый РНК, в этих первых клетках играл важную роль, а некоторые самосплайсирующиеся нитроны сохранились до сих пор, например в ядерных генах рРНК у Telrahymena, у бактериофага Т4 и в некоторых генах митохондрий и хлоропластов. В этих случаях большие фрагменты последовательностей нитронов продемонстрировали крайне высокую консервативность, вероятно, в связи с тем, что для образования каталитической поверхности на молекуле РНК они должны были складываться. Хорошо различимы два класса самосплайсирующихся интронов: нитроны I группы начинают реакцию сплайсинга, связываясь с гуаниновым нуклеотидом в последовательности нитрона, при этом G активируется и образует ударную группу, которая разрывает первую из фосфодиэфирных связей, расщепляемых при сплайсинге (связь на 5'-сайте сплайсинга). У интронов II группы эту роль выполняет адениновый нуклеотид; в результате его действия образуется структура, напоминающая лассо. Иными словами, ход реакции в этих двух случаях одинаков, и оба они, по-видимому, представляют собой составные части существовавших некогда механизмов (рис. 9-87).

Рис. 9-87. Два известных класса самосплайсирующихся интронов. У интронов группы I для инициации сплайсинга с определенным сайтом связывается свободный G-нуклеотид (см. рис. 3-19), а у интронов группы II используется особым образом активированный А-нуклеотид. Эти две группы интронов изображены таким образом, чтобы были видны их общие свойства. В обоих случаях ускорение реакции определяется белками, но в катализе участвует и РНК, входящая в состав интрона. Механизм, используемый нитронами группы II, приводит к образованию лассо-подобных структур и напоминает реакцию, катализируемую сплайсосомой (ср. рис. 9-82). (По Т.К. Cech, Cell 44: 207-210, 1986.)
В ходе эволюции сплайсинга ядерной РНК сохранились реакции, используемые самосплайсирующимися нитронами II группы, однако каталитическая функция последовательностей нитронов перешла к отдельным компонентам сплайсосом. Следовательно, маленькие РНК U1 и U2 вполне могут быть остатками, например, последовательностей катализаторов. Передача функций катализа от интрона к сплайсосоме, вероятно, сняла большинство ограничений с эволюции интронов и сделала возможным появление новых интронных последовательностей.
9.4.15. Экспорт мРНК в цитоплазму происходит только после завершения сплайсинга [55]
Согласно общепринятой гипотезе (гл. 8), зрелые молекулы мРНК узнаются рецепторными белками (входящими в состав ядерных пор), которые способствуют продвижению мРНК в цитоплазму посредством активного переноса. Однако основные белки, входящие в состав гяРНП, и различные процессирующие молекулы, связанные в ядре с РНК, по-видимому, никогда не покидают ядро и, вероятно, соскальзывают с РНК по мере ее продвижения через ядерные поры (рис. 9-88). Изучение мутантов дрожжей показало, что в случае РНК, имеющей сайты сплайсинга, процесс переноса может начаться лишь после завершения сплайсинга. У условно летальных мутантов дрожжей, не способных сплайсировать свою РНК при высокой температуре, все несплайсированные предшественники мРНК остаются в ядре, тогда как мРНК, не требующие сплайсинга (а таких большинство у этого одноклеточного эукариотического организма), нормально переносятся в цитоплазму. Это наблюдение ВПОЛНе согласуется с гипотезой, согласно которой РНК задерживаются связанными с ними сплайсосомами, образующими многочисленные агрегаты внутри ядра высших эукариот. Такие агрегаты могут служить «островками сплайсинга», хотя, как они образуются и функционируют, пока неизвестно (рис. 9-89). Возможно, они представляют собой аналоги ядрышка, значительно большей и заметной структуры в ядре, чья организация и функция изучены хорошо.
Известно, что ядрышко - это место в ядре, где из РНК-предшественника образуются молекулы рибосомной РНК (рРНК), которые связываются с соответствующими белками и собираются в рибосомные частицы. Однако перед тем, как обсуждать строение ядрышка, необходимо рассмотреть синтез молекул-предшественников рРНК на генах рРНК.
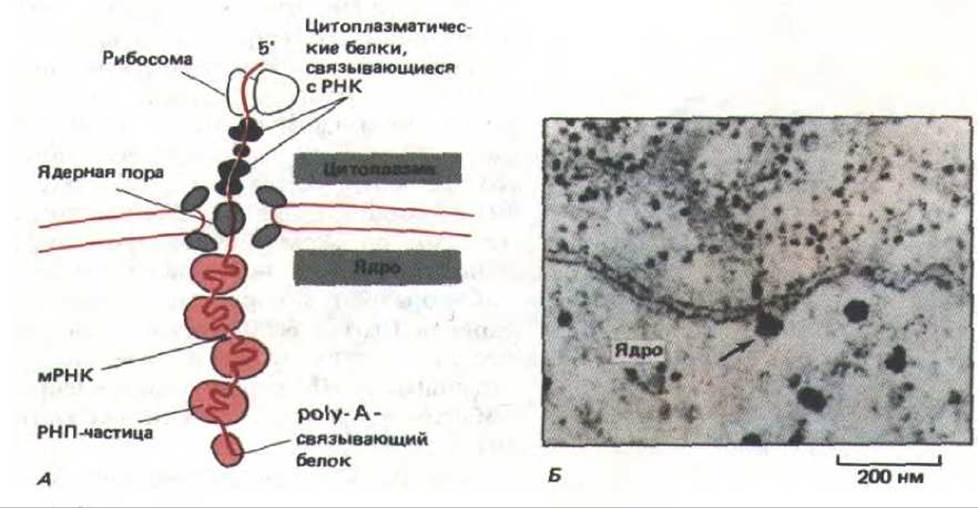
Рис. 9-88. Перемещение молекул мРНК через ядерные поры. А. Изменения, происходящие в белках, связанных с молекулой РНК при ее выходе из ядра. Б. Электронная микрофотография большой молекулы мРНК, образовавшейся в глетке слюнной железы насекомого. Очевидно, эта молекула «поймана» в момент выхода в цитоплазму (указано стрелкой). (По В. J. Stevens and H. Swift, J. Cell Biol. 31: 55-77, 1966.)
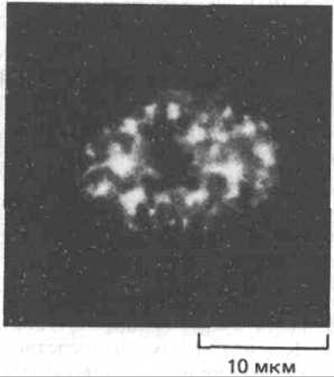
Рис. 9-89. Иммунофлуоресцентное мечение ядра фибробласта человека моноклональными антителами к мяРНП-частицам, участвующим в сплайсинге молекул-предшественников мРНК. Частицы мяРНП образуют большие агрегаты, которые могут функционировать как «островки сплайсинга». Антитела выявляют определенные белки, присутствующие в ряде мяРНК и участвующие в работе сплайсосомы. (С любезного разрешения N. Ringertz.)
10-23
9.4.16. Рибосомные РНК синтезируются на тандемно расположенных копиях идентичных генов [56]
Многие белки, присутствующие в дифференцирующейся клетке в больших количествах (например, гемоглобин в эритроцитах и миоглобин в клетках мышц), синтезируются с генов, представленных в гаплоидном геноме лишь одной копией. Количество этих белков в клетке велико благодаря тому, что с каждой из многих молекул мРНК, транскрибируемых с кодирующих их генов, за минуту может транслироваться до 10 белков. В норме в каждом клеточном поколении это приводит к образованию более 10000 молекул белка на одну молекулу мРНК. Однако такого рода амплификация невозможна в случае рибосомной РНК, так как эти молекулы и являются конечным продуктом гена. Тем не менее, в растущей клетке высших эукариот для того, чтобы получить необходимые 10 млн рибосом за каждую генерацию, должно синтезироваться 10 млн копий каждого типа молекул рибосомной РНК. Такие количества могут образоваться лишь в том случае, если клетка содержит множественные копии генов, кодирующих рибосомную РНК (гены рРНК).
Даже E.coli для обеспечения клетки нужным количеством рибосом должна иметь семь копий гена рРНК. В клетках человека содержится около 200 копий гена рРНК на гаплоидный геном, причем эти копии распределены небольшими кластерами по пяти различным хромосомам, В клетках шпорцевой лягушки Xenopus обнаружено приблизительно 600 копий гена рРНК на одной хромосоме. Множественные копии высококонсервативных генов рРНК в любой хромосоме расположены в виде серии тандемных повторов, отделенных друг от друга нетранскрибируемым участком ДНК, который называется спейсером. В гл. 10 мы изложим современные представления о механизме возникновения таких тандемно расположенных генов.
Благодаря тому, что кластеры тандемно повторяющихся генов рРНК сгруппированы и очень активно транскрибируются, они хорошо видны на электронномикроскопических препаратах хроматина. Молекулы РНК-полимеразы и ассоциированные с ними новообразованные транскрипты (на один ген их приходится до 100 и более) упакованы таким образом, что структура приобретает вид «елочки» (рис. 9-90). Как отмечалось ранее (см. рис. 9-73), верхушка каждой такой елочки представляет собой точку инициации транскрипции; конец же гена рРНК четко определяется по внезапному исчезновению молекул РНК-полиме-разы и соответствующих транскриптов.
Гены рРНК транскрибируются РНК-полимеразой I с образованием идентичных транскриптов. В клетках человека такие первичные транскрипты генов рРНК, известные как 45S-PHK, имеют в длину около 13000 нуклеотидов. Прежде чем покинуть ядро в составе собранной рибосомной частицы, молекула 45S-PHK подвергается специфическому расщеплению, в результате чего образуется по одной копии 28S-PHK (около 5000 нуклеотидов), 18S-PHK (около 2000 нуклеотидов) и 5,88-РНК (около 160 нуклеотидов), которые, собственно, и являются компонентами рибосом. Общее происхождение всех трех типов рРНК из одного и того же первичного транскрипта служит гарантией того, что они образуются в равных количествах. Остальная часть этого транскрипта (около 6000 нуклеотидов) деградирует в ядре (рис. 9-91). Не исключено, что эти «излишние» последовательности молекулы-предшественника рРНК играют определенную роль на ранних этапах сборки рибосомы, происходящих непосредственно по завершении синтеза 45S-РНК.
Другие тандемно повторяющиеся гены, также разделенные нетранслируемыми спейсерами, представлены генами 5S-PHK большой рибосомной субъединицы (это единственная рРНК, которая транскрибируется отдельно от других). Гены 5S-pPHK насчитывают в длину всего лишь около 120 нуклеотидных пар и как большинство других генов, кодирующих маленькие стабильные РНК (наиболее известные из них-это гены тРНК), транскрибируются РНК-полимеразой III. У человека имеется приблизительно 2000 генов 5S-pPHK, которые тандемно расположены внутри единственного кластера, локализованного совершенно в другой области, чем остальные гены рРНК. Остается неизвестным, почему этот тип рРНК транскрибируется отдельно.
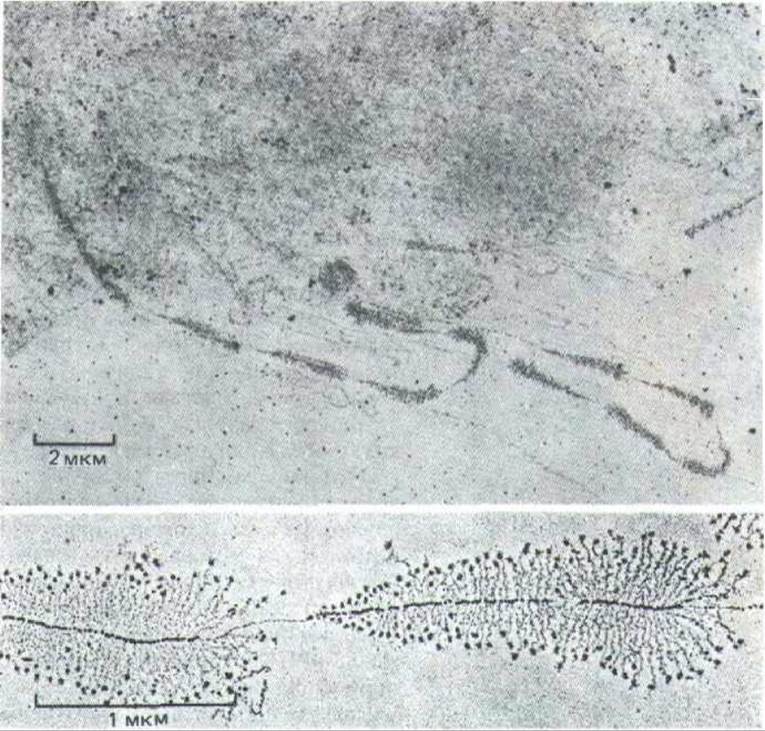
Рис. 9-90. Транскрипция тандемно расположенных генов рРНК, визуализированная с помощью электронного микроскопа. На верхней микрофотографии при меньшем увеличении хорошо различимы чередующиеся транскрибируемые гены и нетранскрибируемые спейсеры. Большие частицы на 5'-конце каждого транскрипта рРНК (нижняя фотография), как полагают, связаны с началом сборки рибосом. Хорошо видны также молекулы РНК-полимераз. (Верхний снимок V. Е. Foe, Cold Spring Harbor Symp. Quant. Biol. 42: 723-740, 1978, нижний снимок-с любезного разрешения Ulrich Scheer.)
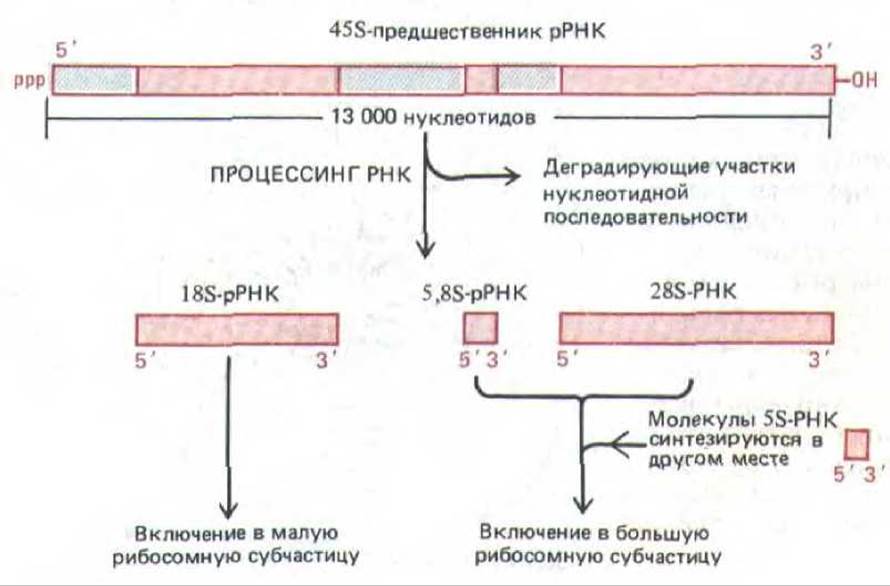
Рис. 9-91. Процессинг 45S-предшественника рибосомных РНК с образованием молекул РНК трех типов. Обратите внимание, что почти половина нуклеотидноЙ последовательности пре-рРНК деградирует в ядре.
9.4.17. Ядрышко-это центр образования рибосом [57]
Непрерывная транскрипция дуплицированных генов обеспечивает клетку достаточным количеством рРНК. Новосинтезированные транскрипты незамедлительно связываются с рибосомными белками с образованием рибосом. Сборка рибосом происходит в ядре, а именно в той его специфической области с диффузной структурой, которая называется ядрышком. В составе ядрышка выявляются большие петли ДНК, содержащие гены рРНК, которые с необычайно высокой скоростью транскрибируются РНК-полимеразой I. Эти петли носят название «ядрышковые организаторы». Самое начало процесса сборки рибосомы можно увидеть в электронный микроскоп: 5'-концы рРНК-транскриптов, составляющих крону «елочек», компактизуются с образованием белок-содержащих гранул (рис. 9-90). Подобные гранулы не образуются на РНК- транскриптах других типов, по-видимому, они отражают первые стадии РНК-белкового взаимодействия, происходящего в ядрышке.
Биосинтетические функции ядрышка можно изучать с помощью кратковременного радиоактивного мечения вновь синтезированной РНК [3Н]-уридином. Через различные промежутки времени, прошедшие со времени включения метки, клеточное содержимое фракционируют и выделяют меченые ядрышки (рис. 9-92). Эти эксперименты показали, что интактные 455-транскрипты сначала образуют крупные комплексы, связываясь с большим количеством различных белков, импортируемых из цитоплазмы, где синтезируются все клеточные белки. Большая часть из 70 различных полипептидных цепей, формирующих рибосому, а также 5S-pPHK включаются именно на этой стадии. Для правильного протекания процесса сборки необходимы и иные молекулы. Например, в ядрышке присутс 3U-мяРНП), которые, как полагают, катализируют сборку рибосом. Эти компоненты остаются в ядрышке, а субъединицы рибосом в готовом виде транспортируются в цитоплазму. Особенно заметным компонентом ядрышка является нуклеолин, хорошо изученный белок, который присутствует в больших количествах, и по-видимому, связывается только с транскриптами рибосомной РНК. Нуклеолин особым образом окрашивается серебром. Такое окрашивание характеризует и все ядрышко в целом.
Во время процессинга 45S-PHK этот гигантский рибонуклеопротеиновый комплекс постепенно теряет часть белков и последовательностей РНК и затем специфически расщепляется, образуя самостоятельные предшественники большой и малой рибосомных субъединиц (рис. 9-93). Через 30 мин после введения радиоактивной метки первые зрелые малые субъединицы рибосом, содержащие меченую 18S-pPHK, выходят из ядрышка и появляются в цитоплазме. Сборка больших рибосомных субъединиц, содержащих 28S, 5,85, и 5S-PHK, требует несколько больше времени (около 1ч), поэтому в ядрышке накапливается гораздо больше недостроенных больших субъединиц, чем малых.
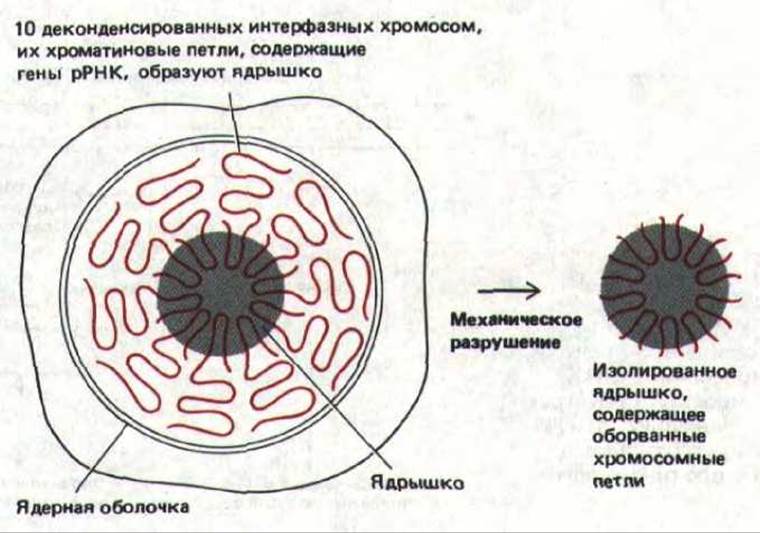
Рис. 9-92. Упрощенная схема клетки человека, иллюстрирующая участие хроматиновых петель, принадлежащих 10 отдельным хромосомам и содержащих гены рРНК, в образовании одного большого ядрышка. Препараты очищенных ядрышек весьма полезны в биохимических исследованиях функций ядрышка; получают такие препараты путем механического отделения образующих ядрышко хроматиновых петель от хромосом, как показано на рисунке.
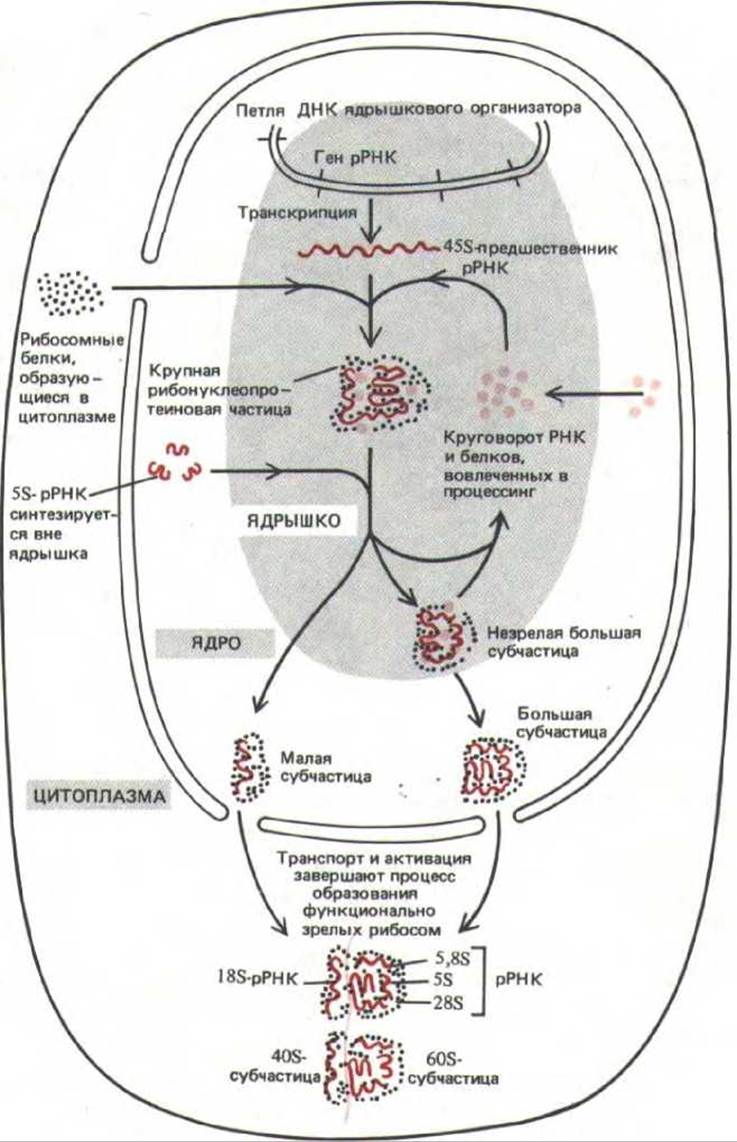
Рис. 9-93. Схема, иллюстрирующая функции ядрышка в биосинтезе рибосом. 458-рРНК-транскрипт упакован в составе крупной рибонуклеопротеиновой частицы, содержащей много рибосомных белков, импортированных из цитоплазмы. В ходе процессинга этой РНП-частицы, протекающего в ядрышке, формируются предшественники большой и малой рибосомных субъединиц; часть гигантского РНП-комплекса при этом деградирует. Предполагается, что окончательное созревание рибосомных субъединиц происходит лишь после того, как они (каждая индивидуально) транспортируются через ядерные поры в цитоплазму.
Заключительные стадии созревания рибосом осуществляются только после выхода рибосомных субъединиц из ядра в цитоплазму. Этим достигается изоляция функционирующих рибосом от незрелых ядерных транскриптов.
9.4.18. Ядрышко-это высокоорганизованная структура внутри ядра [57]
По данным световой микроскопии большое сфероидальное ядрышко-наиболее легко выявляемая структура в ядре немитотической клетки. Неудивительно поэтому, что оно было столь скрупулезно исследовано первыми цитологами: обзор по данной теме, датированной 1898 г., содержал около 700 ссылок. К 1940 г. выяснилось, что в ядрышке находится большое количество РНК и белков, однако его биологическая роль, связанная с синтезом рибосомных РНК и сборкой рибосом, оставалась неизвестной вплоть до 60-х годов.
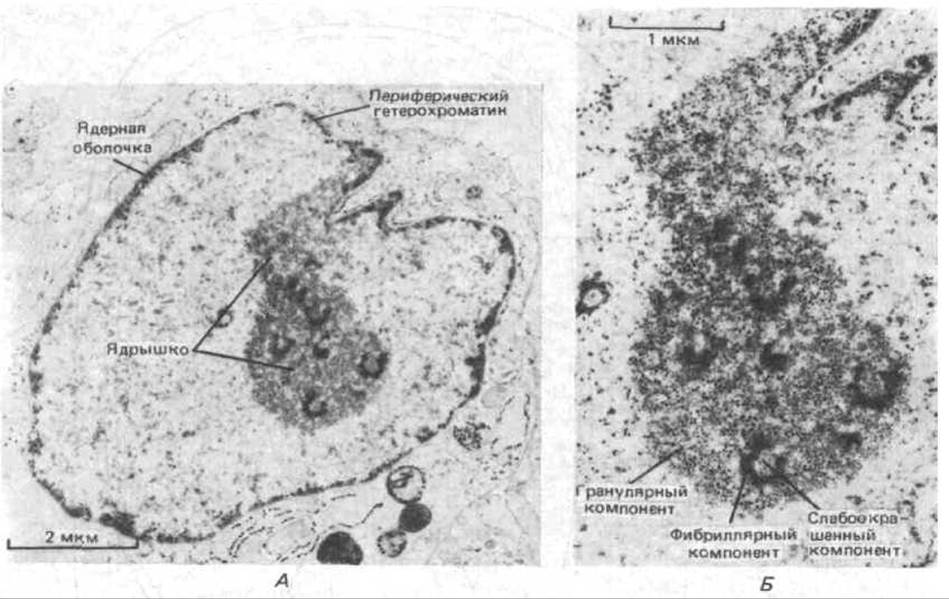
Рис. 9-94. Электронная микрофотография тонкого среза ядрышка в фибробласте человека. Хороню видны три дискретные зоны. А. Общий вид ядрышка. Б. Детализация. (С любезного разрешения Е. G. Jordan and J. McGovern.)
Некоторые детали строения ядрышка можно увидеть с помощью электронной микроскопии. В отличие от цитоплазматических органелл ядрышко не имеет мембраны, которая окружала бы его содержимое. Похоже, что оно образовано недозрелыми предшественниками рибосом, специфически связанными друг с другом неизвестным образом. На типичной электронной микрофотографии ядрышка можно различить три дискретные зоны (рис. 9-94): 1) слабоокрашенный компонент, содержащий ДНК из области ядрышкового организатора хромосомы, 2) гранулярный компонент, в состав которого входят частицы диаметром 15 нм, представляющие наиболее зрелые предшественники рибосомных частиц, и 3) плотный фибриллярный компонент, состоящий из множества тонких (5 нм) рибонуклепротеиновых фибрилл, представляющих собой РНК-транскрипты.
Размер ядрышка отражает степень его функциональной активности, которая широко варьирует в различных клетках и может изменяться в индивидуальной клетке. Так, в некоторых покоящихся клетках растений ядрышко очень мало, тогда как в клетках, продуцирующих большое количество белков, оно может занимать до 25% объема всего ядра. Изменение размеров ядрышка связано главным образом с уменьшением или увеличением доли гранулярного компонента, что, в свою очередь, вероятно, контролируется на уровне транскрипции рибосомных генов: по данным электронной микроскопии доля активных рибосомных генов, равно как и эффективность транскрипции каждого гена, изменяется в зависимости от обстоятельств.
9.4.19. После каждого митоза ядрышко образуется заново из специфических участков хромосомы [58]
Внешний вид ядрышка заметно меняется в зависимости от фазы клеточного цикла. С началом митоза ядрышко уменьшается в размерах, а затем вовсе исчезает по мере того, как происходит конденсация хромосом и прекращается синтез всех типов РНК; как правило, в метафазной клетке ядрышко не обнаруживается. В конце митоза (в телофазе), когда возобновляется синтез рибосомных РНК, миниатюрные ядрышки вновь возникают на участках хромосом, содержащих гены рибосомной РНК (рис. 9-95).
В клетках человека гены рРНК локализуются на концах пяти пар хромосом, т.е. на 10 хромосомах из имеющихся 46 (см. рис. 9-40). Поэтому сразу после митоза в клетке человека образуется 10 маленьких ядрышек; их редко удается увидеть, так как они быстро растут и сливаются друг с другом в одно большое ядрышко, типичное для многих интерфазных клеток (рис. 9-96).
Что же происходит с РНК и белковым компонентом дезагрегировавшего в процессе митоза ядрышка? По-видимому, какая-то часть их распределяется по всем метафазным хромосомам и переносится в ядра дочерних клеток. В телофазе митоза при деконденсации хромосом эти «старые» ядрышковые компоненты могут участвовать в построении новых ядрышек.
9.4.20. Во время интерфазы отдельные хромосомы находятся на определенных местах в ядре [59]
Как отмечалось выше, при образовании ядрышка нужные для этого гены интерфазных хромосом собираются вместе в определенной области ядра. В связи с этим возникает вопрос, а не расположены ли хромосомы в ядре неслучайным образом? Этот важнейший вопрос был впервые поставлен биологами еще в конце XIX в, но до сих пор убедительный ответ на него не получен.
Некоторая упорядоченность в расположении хромосом связана с конфигурацией, которую они имеют в конце митоза. Известно, что непосредственно перед делением клетки конденсированные хромосомы растаскиваются к полюсам микротрубочками, которые прикреплены к центромерам. Таким образом, центромеры идут впереди, а дистальные плечи хромосом, оканчивающиеся теломерами, оказываются позади. Хромосомы во многих ядрах в течение всей интерфазы стремятся сохранить эту так называемую Раблъ-ориентацию, при которой их центромеры обращены к одному полюсу ядра, а теломеры направлены к противоположному полюсу (рис. 9-97, А). В некоторых случаях полюсы ядра определенным образом ориентированы в клетке: например, в ранних зародышах дрозофилы все центромеры направлены к верхушке (рис. 9-97, A). Подобная фиксированная ориентация ядра может в значительной мере влиять на полярность клетки, однако непонятно, какой эксперимент следует провести для проверки этого предположения.
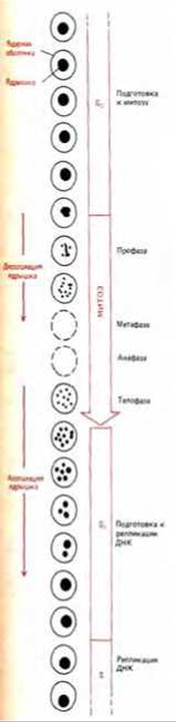
Рис. 9-95. Внешний вид ядрышка в клетках человека изменяется на разных стадиях клеточного цикла. На схеме изображено только клеточное ядро.
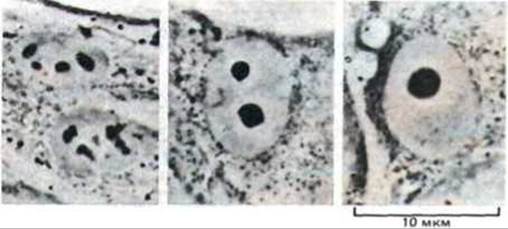
Рис. 9-96. Микрофотографии культивируемых фибробластов человека, демонстрирующие различные стадии ассоциации ядрышка. (С любезного разрешения Е. G. Jordan and J. McGovern.)
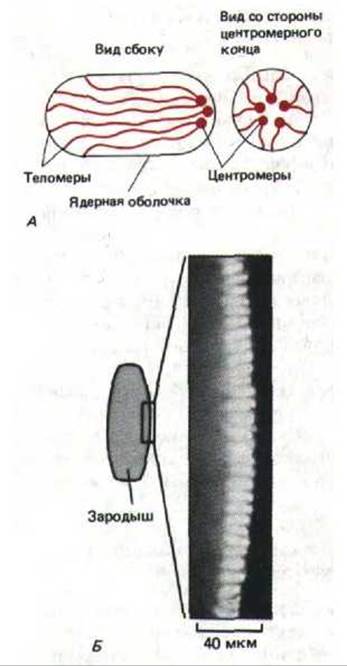
Рис. 9-97. Поляризованная ориентация хромосом в интерфазных клетках раннего эмбриона дрозофилы. А. Рабль-ориентация: все центромеры направлены к одному полюсу ядра, а все теломеры-другому. Обратите внимание, что у эмбриона все ядра имеют удлиненную форму. Б. Полученная при небольшом увеличения световая микрофотография эмбриона дрозофилы на стадии клеток бластодермы; хромосомы окрашены флуоресцирующим красителем. Следует отметить, что области, окрашенные ярче всего (хромоцентры), ориентированы к наружной поверхности зародыша, и, следовательно, обращены в сторону апикальной плазматической мембраны каждой клетки. (С любезного разрешения John Sedat.)
В большинстве интерфазных клеток различные хромосомы невозможно отличить друг от друга, и, следовательно, трудно определить их расположение точнее, чем это описано выше. Исключение составляют гигантские политенные хромосомы интерфазных клеток личинок дрозофилы. На этих хромосомах отдельные диски различаются достаточно хорошо, чтобы в интактном ядре можно было точно определить положение конкретных генов. Исследования показали, что набор интерфазных хромосом в целом не характеризует высокая упорядоченность, хотя Рабль-ориентация сохраняется. В двух на вид идентичных клетках по соседству часто расположены разные хромосомы.
Анализ политенных хромосом свидетельствует о том, что в интерфазном ядре каждая хромосома расположена отдельно, т.е. разные хромосомы не сильно перевиты друг с другом (рис. 9-98). Более того, установлено, что в интерфазном ядре и другие типы хромосом, а не только политенные, стремятся занять дискретные домены. Например, методом гибридизации in situ с соответствующими ДНК-зондами можно выявить отдельную хромосому в гибридных млекопитающих, растущих в культуре (рис. 9-99). Замечено, что большая часть ДНК такой хромосомы занимает очень небольшую часть интерфазного ядра. Этот факт свидетельствует о том, что каждая отдельная хромосома остается компактной и структурированной, благодаря чему отдельные ее части могут активно участвовать в синтезе РНК.

Рис. 9-98. Стереоизображение политенных хромосом в отдельном ядре клетки слюнных желез дрозофилы. Большой шар-это ядрышко. Тело-меры обычно располагаются на поверхности ядерной оболочки напротив той его области, под которой расположено ядрышко; вблизи ядрышка расположены все центромеры. Хромосомы в ядре никогда не перепутываются, однако детали их укладки и соседи в одинаковых ядрах различаются. (С любезного разрешения Mark Hoch-strasser and John W. Sedat.)
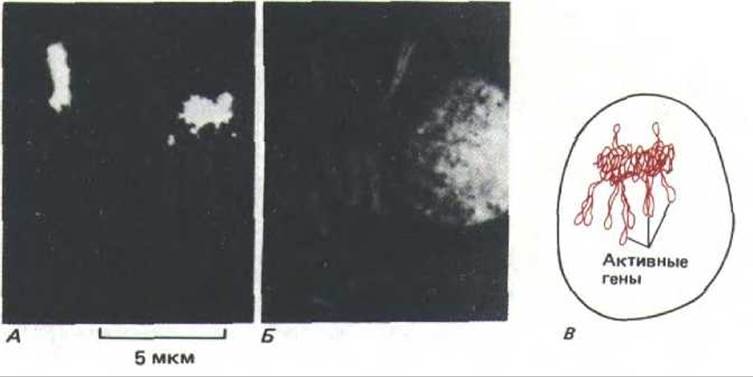
Рис. 9-99. Селективное мечение отдельной хромосомы в ядре культивируемых клеток млекопитающих в интерфазе. А. Результаты гибридизации in situ (с использованием флуоресцирующего зонда), позволяющей идентифицировать отдельные хромосомы человека в гибридной линии клеток человек - хомяк. Б. Тот же препарат, вся ДНК которого помечена флуоресцирующей меткой. В. Схема хромосомы человека в интерфазном ядре, представленном в части А. (А и Б-с любезного разрешения Joyce A. Kobori and David R. Cox.)
9.4.21. Насколько сильно структурировано ядро? [60]
Внутренняя часть ядра - это не случайно перемешанные молекулы РНК, ДНК и белков, входящих в его состав. Выше уже шла речь о том, что ядрышко представляет собой эффективное устройство для сборки рибосом, а кластеры сплайсосом, по-видимому, организованы в виде дискретных островков, где и происходит сплайсинг РНК (см. рис. 9-89). Упорядоченность структур хорошо видна и на электронных микрофотографиях ядерных пор: хроматин расположен вдоль внутренней ядерной мембраны, но отсутствует вокруг каждой ядерной поры и под ней (т. е. проход между цитоплазмой и нуклеоплазмой свободен (рис. 9-100). Более того, оказалось, что в некоторых случаях положение ядерных пор на ядерной оболочке не случайно, а строгим образом упорядочено (рис. 9-101). Подобная упорядоченность свидетельствует о соответствующей организации ядерной ламины, к которой и прикреплены поры.
Существует ли какая-либо внутриядерная структура, аналогичная цитоскелету, на которой и расположены ядерные компоненты? Многие биологи, изучающие клетку, убеждены, что такая структура существует. Ядерным матриксом или скелетом называют нерастворимый материал, остающийся в ядре после ряда биохимических экстракций. Можно показать, что белки, входящие в его состав, связывают определенные последовательности ДНК, называемые SAR или MAR (области, связанные со скелетом - scaffold -associated regions ипи с мятриксом - matrix-associated regions). Предполагают, что такие последовательности образуют основание петли ДНК (см. рис. 9-34). Благодаря сайтам прикрепления хромосом матрикс может участвовать в организации хромосом, определять локализацию генов, регулировать транскрипцию и репликацию ДНК в ядре. Однако в связи с тем, что структурные компоненты матрикса еще не идентифицированы, остается неизвестным, представляет ли собой матрикс, полученный в ходе экспериментов, структуру, идентичную той, которая присуща интактным клеткам.
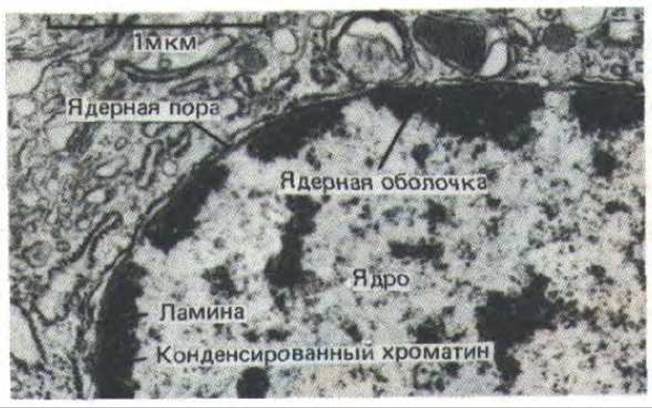
Рис. 9-100. Электронная микрофотография ядра клетки млекопитающего. Отчетливо видно, что конденсированный хроматин, выстилающий ядерную оболочку, отсутствует вокруг ядерных пор. (С любезного разрешения Zarry Gerace.)

Рис. 9-101. Электронная микрофотография ядерной оболочки споры папоротника, полученная с помощью метода «замораживание - скалывание». Видно упорядоченное расположение комплексов ядерных пор, лежащих параллельными рядами. На ядерной оболочке других клеток наблюдались либо сконцентрированные в одной области кластеры ядерных пор, либо необычные зоны, лишенные ядерных пор (они определенным образом ориентированы относительно других структур клетки). (С любезного разрешения Don H. Northcote, по К. Roberts and D. H. Northcote, Microsc. Acta 71: 102-120, 1971.)
Заключение
РНК-полимераза, фермент, катализирующий транскрипцию ДНК, представляет собой сложную молекулу, состоящую из многих полипептидных цепей. В эукариотических клетках обнаружено три РНК-полимеразы I, II и III. Эти ферменты эволюционно связаны друг с другом и с бактериальной РНК-полимеразой, у них имеются одинаковые субъединицы. По-видимому, после инициации транскрипции от каждого фермента отделяются одна или несколько субъединиц, называемых факторами инициации. Вместо них к ферментам присоединяются субъединицы, называемые факторами элонгации. Они необходимы для удлинения цепи РНК, ее терминации и модификации. Вероятно, факторы элонгации у различных типов полимераз разные, именно этим можно объяснить, почему транскрипты, синтезируемые каждым ферментом, модифицируются по-разному.
Большая часть мРНК клетки образуется в результате сложного процесса, начинающегося с синтеза гетерогенной ядерной РНК. Первичные транскрипты гяРНК синтезируются РНК-полимеразой II. Эти транскрипты затем кэпируются путем добавления к 5'-концу специального нуклеотида и после этого полиаденилируются на 3'-конце. Модифицированные молекулы РНК обычно затем подвергаются процессингу, в ходе которого из середины гяРНК удаляются последовательности интронов. Эта реакция катализируется большим рибонуклепротеиновым комплексом, известным под названием сплайсосома. При этом значительная часть первичного транскрипта РНК удаляется и распадается в ядре. Хотя обычно гяРНК составляет около половины всей РНК, синтезированной клеткой, образующаяся в результате процессинга мРНК,-это всего лишь 3% РНК клетки.
В отличие от генов, кодирующих белки и транскрибирующихся полимеразой U, гены, кодирующие большую часть структурных РНК, транскрибируются полимеразами I и Ш. Эти гены обычно амплифицированы, собраны в тандемные кластеры. РНК-полимераза III синтезирует стабильные небольшие РНК, включая тРНК и 5S-pPHK. РНК-полимераза I ответственна за образование большой молекулы-предшественника рРНК (45S-pPHK). Все рибосоми клетки, за исключением митохондриальных и хлоропластных, собираются в ядрышке-отдельной ядерной органелле, формирующейся вокруг тандемно расположенных генов рРНК, которые собираются вместе из нескольких хромосом.
Литература
Общая
Lewin В. Gene expression, Vol. 2. Eucaryotic Chromosomes, 2nd ed. New York, Wiley, 1980.
Levfin B. Genes, 3rd ed. New York, Wiley, 1987.
Newport J. W., Forbes D. J. The nucleus: structure, function, and dynamics. Annu. Rev. Biochem, 56, 535--566, 1987.
Watson J. D., Hopkins N. H.. Roberts J. W., Sleitz J. A., Weiner A. M. Molecular Biology of the Gene, 4th ed. Menlo Park, CA, Benjamin-Cummings, 1987.
Цитированная
1. Adolph К. W., ed. Chromosomes and Chromatin, Vols. 1-3. Boca Raton FL, CRC Press, 1988.
Felsenfeld G. DNA. Sci. Am., 253 (4), 58-67, 1985.
Hsu T.C. Human and Mammalian Cytogenetics:A Historical Perspective. New York, Springer-Verlag, 1979.
2. Kacenoff R, KlotzL. C., Zimm В. Н. On the nature of chromosome-sized DNA molecules. Cold Spring Harbor Symp. Quant. Biol., 38, 1-8, 1974.
3. Burke D. Т., Carle G. F., Olson M. V. Cloning of large segments of exogenous DNA into yeast by means of artificial chromosome vectors. Science, 236, 806-812, 1987.
Murray A. W. Chromosome structure and behavior. Trends Biochem. Sci., 10, 112-115, 1985.
4. Gall J. G. Chromosome structure and the C-value paradox. J. Cell Biol., 91, 3s-14s,1981.
Ohta Т., Kimura M. Functional organization of genetic material as a product of molecular evolution. Nature, 233, 118-119, 1971.
5. Mapping and Sequencing the Human Genome. Washington DC, National Academy Press, 1988.
Wilson A. C., Ochman H., Prager Е. М., Molecular time scale for evolution. Trends Genet., 3, 241-247, 1987.
6. Fried, M., Crothers D. M. Equilibria and kinetics of lae-repressor-operator interaction by polyacrylamide gel electrophoresis. Nucleic Acids Res., 9, 6505-6525, 1981.
7. Kadonaga J. Т., TjianR. Affinity purification of sequence-specific DNA binding proteins. Proc. Natl. Acad. Sci. USA, 83, 5889-5893, 1986. Rosenfeld P.J., Kellv T.J. Purification of nuclear factor I by DNA recognition site affinity chromafography. J. Biol. Chem., 261, 1398-1408, 1986.
Staudt L. M. et al. Cloning of a lymphoid-specific cDNA encoding a protein binding the regulatory octamer DNA motif. Science, 241, 577-580, 1988.
8. Pabo С. Т., Sauer R. T. Protein-DNA interactions. Annu. Rev. Biochem., 53, 293-321, 1984. Schief R. DNA binding by proteins. Science, 241, 1182 1187, 1988.
9. Ptashne M. A. Genetic Switch: Gene Control and Phage Lambda. Palo Alto CA, Black well, 1986.
Takeda Y, Ohlendorf D.H., Anderson, W.F., Matthews R. W. DNA-binding proteins. Science, 221, 1020-1026, 1983.
10. Berg 0. G., van Hippel P. H. Selection of DNA binding sites by regulatory proteins. Trends Biochem. Sci., 13, 207-211, 1988.
von Hippel P.H., Bear D.G., Morgan W.D., McSwiggen J.A., Protein-nucleic acid interaction in transcription. Annu. Rev. Biochem., 53, 389446, 1984.
11. Dickerson R.E. The DNA helix and how it is read. Sci. Am., 249(6). 94-111, 1983.
Drew H.R., McCallM.J., Callandine C.R. Recent studies of DNA in the crystal. Annu. Rev. Cell Biol,. 4, 1-20, 1988.
12. Koo H. S., Crothers D. M. Calibration of DNA curvature and a unified description of sequence-directed bending. Proc. Natl. Acad. Sci. USA, 85, 1763-1767, 1988.
Lilley D. Bent molecules-how and why? Nature, 320, 487, 1986.
Marini J. C., Levene S. D., Crothers D. M., Englund P. T. Bent helical structure in kinetoplast DNA. Proc. Natl. Acad. Sci. USA, 80, 76787682, 1982.
13. EcholsH. Multiple DNA-protein interactions governing high-precision DNA transactions. Science, 233, 1050-1056, 1986.
Thompson J. F., de Vargas L. M., Koch C., Kahmann R., Landy A. Cellular factors couple recombination with growth phase: characterization of a new component in the lambda site-specific recombination pathway. Cell, 50, 901 -908, 1987.
14. Isenberg I. Histones. Annu. Rev. Biochem., 48, 159-191, 1979.
van Holt C. Histones in perspective. Bioessays, 3, 120-124, 1985.
Wells D. E. Compilation analysis of histones and histone genes. Nucleic Acids Res., 14, rll9-rl49, 1986.
Wu R. S., Panusz H. Т., Hatch C. L., Banner W. M. Histones and their modifications. CRC Crit. Rev. Biochem., 20, 201-263, 1986.
15. Chromatin. Cold Spring Harbor Symp. Quant. Biol., Vol. 42, 1978.
Kornberg R.D., Klug A. The nucleosome. Sci. Am., 244 (2), 52-64, 1981.
McGhee, J.D., Felsenfeld G. Nucleosome structure. Annu. Rev. Biochem., 49, 1115-1156, 1980.
Richmond T.J., Finch J.T., Rushton В., Rhodes D., Klug A. Structure of the nucleosome core particle at 7 A resolution. Nature, 311, 532-537, 1984.
16. Simpson R. T. Nucleosome positioning in vivo and in vitro. Bioessays, 4, 172-176, 1986. Trovers A. A. DNA bending and nucleosome positioning. Trends Biochem. Sci., 12, 108-112, 1987.
17. Eissenberg J.C., Cartwright I.L., Thomas G.H., Elgin S.C. Selected topics in chromatin structure. Annu. Rev. Genet., 19, 485-536, 1985.
Emerson B. M. Lewis C. D., Felsenfeld G. Interaction of specific nuclear factors with the nuclease-hypersensitive region of the chicken adult p-globin gene: nature of the binding domain. Cell 41, 21-30, 1985.
Gross D. S., Garrad W. T. Nuclease hypersensitive sites in chromatin. Annu. Rev. Biochem., 57, 159-198, 1988.
18. Belmont A.S., Sedat J.W., Agard D.A. A three-dimensional approach to mitotic chromosome structure: evidence for a complex hierarchial organization. J. Cell Biol., 105, 77-92, 1987.
Pederson D.S., Thoma F., Simpson R. Core particle, fiber, and transcriptionally active chromatine structure. Annu. Rev. Cell Biol., 2, 117-147, 1986.
19. Allan J., Hartman P. G., Crane-Robinson C., Aviles F. X. The structure ofhistone HI and its location in chromatin. Nature, 288, 675-679, 1980.
Clark D.J., Thomas J.O. Salt-dependent cooperative interaction ofhistone HI with linear DNA. J. Мої. Biol., 187, 569-580, 1986.
Coles L.S., Robins A.J., Madley L.K. Wells J.R. Characterization of the chiken histone HI gene complement: generation of a complete set of vertebrate HI protein sequences. J. Biol. Chem, 262, 9656 9663, 1987.
Thoma F., Koller Т., Klug A. Involvement of histone HI in the organization of the nucleosome and of the salt-dependent superstructures of chromatin. J. Cell Biol., 83, 403-427, 1979.
20. De Bernardin W., Koller Т., Sogo J. M. Structure of in vivo transcribing chromatin as studied in simian virus 40 minichromosomes. J. Мої. Biol. 191, 469-482, 1986.
Losa R., Brown D.D. A bacteriphage RNA polymerase in vitro through a nucleosome core without displacing it. Cell, 50, 801-808, 1987.
21. Benyajati C., Worcel A. Isolation, characterization, and structure of the folded interphase genome of Drosophila melanogaster. Cell, 9, 393-408, 1976.
Gasser S. M., Laemmli U. K. A glimpse at chromosomal order. Trends Genet., 3, 16-22, 1987.
SchmidM. B. Structure and funcfion ofthe bacterial chromosome. Trends Biochem. Sci., 13, 131-135, 1988.
22. Marsden M., Laemmli U. K. Metaphase chromosome structure: evidence for a radial loop model. Cell, 17, 849-858, 1979.
Georgiev G.P., Nedospasov S.A., Bakaev V. V. Supranucleosomal levels of chromatin organization. In: The Cell Nucleus (H. Busch, ed.), Vol. 6, pp. 3-34. New York, Academic Press, 1978.
23. Holmquist G. DNA sequences in G-bands and R-bands. In: Chromosomes and Chromatin Structure (K.W. Adolph, ed.), Vol. 2, pp. 75-122. Boca Raton, FL, CRC Press, 1988.
Lewin B. Gene Expression, Vol. 2. Eucaryotic Chromosomes, 2nd ed., pp. 428-440, New York, Wiley, 1980.
24. Bostock C. J., Sumner A. T. The Eucaryotic Chromosome, pp. 347-374. Amsterdam, North-Holland, 1978. Callan H.G. Lampbrush chromosomes. Proc. R. Soc. Lond. (Biol.), 214, 417-448, 1982.
Roth M.B., Gall J.G. Monoclonal antibodies that recognize transcription unit proteins on newt lampbrush chromosomes. J. Cell Biol., 105, 1047-1054, 1987.
25. Agard D. A., Sedat J. W. Three dimensional architecture of a polytene nucleus.Nature, 302, 676-681, 1983.
Beermann W. Chromosomes and genes. In: Developmental Studies on Giant Chromosomes (W. Beermann, ed.), pp. 1-33. New York, Springer-Verlag, 1972.
26. Ashburner M., Chihara C., Meltzer P., Richards G. Temporal control of puffing activity in polytene chromosomes. Cold Spring Harbor Symp.
Quant. Biol, 38, 655-662, 1974.
Lamb M. M., Daneholt В., Characterization of active transcription units in Balbiani rings of Chironomus tentans. Cell, 17, 835-848, 1979.
27. Bossy В., Hall L.M., Spierer P. Genetic activity along 315 kb of the Drosophilachromosome. EMBO J., 3, 2537-2541, 1984.
Hill R.J., Rudkin G. Polytene chromosomes: the status of the band-interband question. Bioessays, 7, 35-40, 1987.
Judd B. H., Young M. W. An examination of the one cistron: one chromomere concept. Cold Spring Harbor Symp. Quant. Biol. 38, 573-579, 1974.
28. Garel A., Zolan M., Axel R. Genes transcribed at divers rates have a similar conformation in chromatin. Proc. Natl. Acad. Sci. USA, 74, 4867 4871, 1977.
Weintraub H., Groudine M. Chromosomal subunits in active genes have an altered conformation. Science, 193, 848-856, 1976.
Yaniv M., Cereghini S. Structure of transcriptionally active chromatin. CRC Crit. Rev. Biochem. 21, 1-26, 1986.
29. Allis C.D. et al. hvl is an evolutionary conserved H2A variant that is preferentially associated with active genes. J. Biol. Chem., 261, 1941-1948, 1986.
Dorbic Т., Wittig В. Chromatin from transcribed genes contains HMG17 only downstream from the starting point of transcription. EMBO J., 6, 2393-2399, 1987.
Hebbes T.R., Thorne A. W., Crane-Robinson C. A direct link between core histone acetylation and transcriptionally active chromatin. EMBO J., 7, 1395-1402, 1988.
Rose S. M., Garrard W. T. Differentiation-dependent chromatin alterations precede and accompany transcription of immunoglobulin light chain genes. J. Biol. Chem., 259, 8534-8544, 1984.
30. Brown S. W. Heterochromatin. Science, 151, 417-425, 1966.
James T. C., Elgin S. C. R. Identification of a nonhistone chromosomal protein associated with heterochromatin in Drosophila melanogaster and its gene. Mol. Cell Biol., 6, 3862-3872, 1986.
Pimpinelli S., Bonaccorsi S., Gatti M., Sandier L. The peculiar genetic organization of Drosophila heterochromatin. Trends Genet., 2, 17-20, 1986.
31. Hand R. Eucaryotic DNA: organization of the genome for replication. Cell, 15, 317-325, 1978.
Huberman J. A., Riggs A. D. On the mechanism of DNA replication in mammalian chromosomes. J. Моl. Biol., 32, 327-341, 1968.
32. Campbell J. Eucaryotic DNA replication: yeast bares its ARSs. Trends Biochem. Sci., 13, 212-217, 1988.
Palzkill T. G., Newlon C. S. A yeast replication origin consists of multiple copies of a small conserved sequence. Cell, 53, 441-450, 1988.
Struhl K., Stinchcomb D. Т., Sherer S., Davis R. W. High-frequency transformation of yeast: autonomous replication of hybrid DNA molecules. Proc. Natl. Acad. Sci. USA, 71, 1035-1039, 1979.
33. Dodson M., Dean F. В., Bullock P., Echols H, Hurwitz J. Unwinding of duplex DNA from the SV40 origin of replication by T antigen. Science, 238, 964-967, 1987.
Li J. J., Kelly T.J. Simian virus 40 DNA replication in vitro. Proc. Natl. Acad. Sci. USA, 81, 6973-6977, 1984.
So A. G., Downey K. M. Mammalian DNA polymerases alpha and delta: current status in DNA replication. Biochemistry, 27, 4591-4595, 1988. Stahl H, Droge P., Knippers R. DNA helicase activity of SV40 large T antigen. EMBO J. 5, 1939-1944, 1986.
34. Russev G., Hancock R. Assembly of new histones into nucleosomes and their distribution in replicating chromatin. Proc. Natl. Acad Sci. USA, 79, 3143-3147, 1982.
Sariban E.R., Wu R.S., Erickson L.C., Banner W.M. Interrelationships of protein and DNA synthesis during replication in mammalian cells. Мої. Cell. Biol. 5, 1279-1286, 1985.
Weintraub H., Worcel A., Alberts B.M. A model for chromatin based upon two symmetrically paired half-nucleosomes. Cell, 9, 409-417, 1976. Worcel A., Han S., Wong M. L. Assembly of newly replicated chromatin. Cell, 15, 969-977, 1978.
35. Blackburn E. H., Szostak J. W. The molecular structure of centromeres and telomeres. Annu. Rev. Biochem., 53, 163-194, 1984.
Greider C. W., Blackburn E. H. The telomere terminal transferase of Tetrahymena is a ribonucleoprotein enzyme with two kinds of primer specificity. Cell, 51, 887-898, 1987.
36. Stubblejield E. Analysis of the replication pattern of Chiese hamster chromosomes using 5-bromodeoxyuridine supression of 33285 Hoechst fluorescence. Chromo-soma, 53, 209-221, 1975.
37. Lima-de-Faria A., JaworskaH. Late DNA synthesis in heterochromatin. Nature, 217, 138-142, 1968.
38. Brown E. H., et al. Rate of replication of the murine immunoglobulin heavy-chain locus: evidence that the region is part of a single replicon.
Мої. Cell. Biol., 7, 450-457, 1987.
39. Ca/lan H. G. DNA Replications in the chromosomes of eukaryotes. Cold Sprind Harbor Symp. Quant. Biol., 38, 195-203, 1974.
Kriegstein H.J., Hogness D.S. Mechanism of DNA replication in Drosophila chromosomes: structure of replication forks and evidence for bidirectionality. Proc. Natl. Acad. Sci. USA, 71, 135-139, 1974.
Mechali M., Kearsey S. Lack of specific sequence requirement for DNA replication in Xenopus eggs compared with high sequence specifity in yeast. Cell, 38, 55-64, 1984.
40. Blow J. J., Laskey R. A. A role for the nuclear envelope in controlling DNA replication within the cell cycle. Nature, 332, 546-548, 1998.
Harland R. Initiation of DNA replication in eukaryotic chromosomes. Trends Biochem. Sci. 6, 71-74, 1981.
Rao P. N.. Johnson R. T. Mammalian cell fusion: studies on the regulation of DNA synthesis and mitosis. Nature, 225, 159-164, 1970.
41. Watson J.D., Hopkins N.H., Roberts J.W., Steitz J.A., Weiner AM. Molecular Biology of the Gene, 4th ed. Menlo Park, CA, Benjamin-Cummings, 1987. (Chapters 13, 20 and 21).
42. Chamberlin M. Bacterial DNA-dependent RNA polymerases. In: The Enzymes, 3rd ed. (P. Boyer, ed.), Vol. 15B, pp. 61-108, New York, Academic Press, 1982.
Greenblatt J., Li J. Interaction of the sigma factor and the nusA gene protein of E.coli with RNA polymerase in the initiation-termination cycle of transcription Cell, 24, 421-428, 1981.
McClure W. Mechanism and control of transcription initiation in prokaryotes. Annu. Rev. Biochem., 54, 171-204, 1985.
Yager T.D., van Hlppel P.H. Transcript elongation and termination in E.coli. In: Escherichia coll and Salmonella typhimurium: Cellular and Molecular Biology (F.C. Neidhardt, ed.), pp. 1241-1275. Washington DC, American Society for Microbiology, 1987.
43. Chambon P. Eucaryotic nuclear RNA polymerases. Annu. Rev. Biochem., 44, 613-638, 1975.
Geiduschek E. P., Tocchim Valentini G.P. Transcription by RNA polymerase III. Annu. Rev. Biochem., 57, 873-914, 1988.
Sentenac A. Eucaryotic RNA polymerases. CRC Crit. Rev. Biochem., 18, 31-91, 1985.
Sollner-Webb В., Tower J. Transcription of cloned eucaryotic ribosomal RNA genes. Annu. Rev. Biochem, 55, 801-830, 1986.
44. Brown D. D. The role of stable complexes that repress and activate eucaryotic genes. Cell, 37, 359-365, 1984.
Workman J. L., Roeder R. G., Binding of transcription factor TFIID to the major late promotor during in vitro nucleosome assembly potentiates subsequent initiation by RNA polymerase II. Cell, 51, 613-622, 1987.
45. Foe V. E., Wilkinson L. E., Laird C. D. Comparative organization of active transcription units in Oncopeltus fasciatus. Cell, 9, 131 146, 1976.
Hastie N.D., Bishop J.O. The expression of three abundance classes of mRNA in mouse tissues. Cell, 9, 761-774, 1976.
Lewin B. Gene Expression, Vol. 2. Eucaryotic Chromosomes, 2nd ed., pp. 708-719. New York, Wiley, 1980.
Miller O. L. The nucleolus, chromosomes, and visualization of genetic activity. J. Cell Biol. 91, 15s-27s, 1981. (A review.)
46. Birnstiel M. L., Busslinger M., Struh K. Transcription termination and 3' processing: the end is in site! Cell, 41, 349-359, 1985.
Friedman D.I., Imperiale M.J., Adhya S.L. RNA 3' end formation in the control of gene expression. Annu. Rev. Genet., 21, 453-488, 1987. Nevins J. R. The pathway of eukaryotic mRNA formation. Annu. Rev. Biochem. 52, 441-466, 1983.
Takagaki Y., Ryner L. C., Manley J. L. Separation and characterization of a poly(A) polymerase and a cleavage/specificity factor required for pre-mRNA polyadenylation. Cell, 52, 731-742, 1988.
47. Sisodia S.S., Sollner-Webb В., Cleveland D.W. Specificity of RNA maturation pathways: RNAs transcribed by RNA polymerase III are not substrates for splicing or polyadenylation. Mol. Cell.. Biol., 7, 3602-3612, 1987.
Smale S. Т., Tjian R. Transcription of herpes simplex virus tk sequences under the control of wild-type and mutant human RNA polymerase I promoters. Mol. Cell. Biol. 5, 352-362, 1985.
48. Chambon P. Split genes. Sci. Am. 244(5), 60-71, 1981.
Crick F. Split genes and RNA splicing. Science, 204, 264-271, 1979.
Darnell J. E., Jr. Variety in the level of gene control in eucaryotic cells. Nature, 297, 365-371, 1982. Perry R. P. RNA processing comes of age. J. Cell Biol., 91, 28s-38s, 1981. (Includes a historical review.)
49. Guthrie C., Patterson B. Splice-ossomal snRNAs. Annu. Rev. Genet., 22, 387-419, 1988.
Dreyfuss G., Swanson M.S., Pinol-Roma S. Heterogeneous nuclear ribonucleoprotein particles and the pathway of mRNA formation. Trends Biochem. Sci. 13, 86-91, 1988.
Osheim Y. N.. Miller O. L., Beyer A. L. RNP particles at splice junction sequences on Drosophila chorion transcripts. Cell, 43, 143-151, 1985. Samarina O.P., Krichevskaya A.A., Georgiev G.P. Nuclear ribonucleoprotein particles containing messenger ribonucleic acid. Nature, 210, 1319-1322, 1966.
Steitz J.A., "Snurps". Sci. Am. 258 (6). 56-63, 1988.
50. Edmonds M. Branched RNA. Bioessays, 6, 22-216, 1987.
Maniatis Т., Reed R. The role of small nuclear ribonucleoprotein particles in pre-mRNA splicing. Nature, 325, 673-678, 1987.
Padgett R. A., Grabowski P. J., Konarska M. M., Seller S., Sharp P. A. Splicing of mRNA precursors. Annu. Rev. Biochem. 55, 1119-1150, 1986.
51. Aebi M., Weissman C. Precision and orderliness in splicing. Trends Genet., 3, 102-107, 1987.
52. Andreadis A., College M.E., Nadal-Ginard B. Generation of protein isoform diversity by alternative splicing: mechanistic and biological implications. Annu. Rev. Cell Biol., 3, 207-242, 1987.
53. Orkin S. H., Kazazian H. H. The mutation and polymorphism of the human p-globin gene and its surrounding DNA. Annu. Rev. Genet., 18, 131-171, 1984.
54. Cech T. R. The generality of self-splicing RNA: relationship to nuclear RNA splicing. Cell, 44, 207-210, 1986.
Ringertz N. et al. Computer analysis of the distribution of nuclear antigenes: studies on the spatial and functional organization of the interphase nucleus. J. Cell Sci. Suppl. 4, 11-28, 1986.
Warner J. Applying genetics to the splicing problem. Genes Dev., 1, 1-3, 1987.
56. Long E. О., Dawid I. B. Repeated genes in eucaryotes. Annu. Rev. Biochem., 49, 727-764, 1980.
Miller O. L. The nucleolus, chromosomes and visualization of genetic activity. J. Cell Biol., 91, 15s-27s, 1981.
57. Hadjiolov A. A. The Nucleolus and Ribosome Biogenesis. New York, Springer-Verlag, 1985.
Jordan E. G., Cullis C. A., eds. The Nucleolus. Cambridge, U. K., Cambridge University Press, 1982.
Sommerville J. Nucleolar structure and ribosome biogenesis. Trends Biochem. Sci., 11, 438-442, 1986.
58. Anastassova-Kristeva M. The nucleolar cycle in man. J. Cell Sci., 25, 103, 1977.
McClintock B. The relation of a particular chromosomal element to the develope-ment of the nucleoli in Zea Mays. Z. Zellforsch. Mikrosk. Anat., 21, 294-323, 1934.
59. ComingsD.E. Arrangement of chromatin in the nucleus. Hum. Genet., 53, 131-143, 1980.
Cremer T. et al. Rabl's model of the interphase chromosome arrangement tested in Chinese hamster cells by premature chromosome condensation and laser-UV-microbeam experiments. Hum. Genet., 60, 46-56, 1980.
Hochstrasser M., Sedat J. W. Three-dimensional organization of Drosophila melano-gaster interphase nuclei. II. Chromosome spatial organization and gene regulation. J. Cell Biol., 104, 1471-1483, 1987.
Manuelidis L. Individual interphase chromosome domains revealed by in situ hubridization. Hum. Genet., 71, 288-293, 1985.
60. Gasser S. M., Laemmli U. K. A glimpse at chromosome order. Trends Genet., 3, 16-22, 1987.
Gerace L., Burke B. Functional organization of the nuclear envelope. Annu. Rev. Cell Biol., 4, 335-374, 1988.
Newport J. W., Forbes D. J. The nucleus: structure, function, and dynamics. Annu. Rev. Biochem., 56, 535-566, 1987.