Основы молекулярной биологии. Часть 2: Молекулярные генетические механизмы - А.Н. Огурцов 2011
Репликация и репарация ДНК
Репликационная вилка
Для того чтобы дуплекс ДНК мог работать как матрица в процессе репликации, две его нити должны быть расплетены (или ещё говорят, расплавлены, melted), чтобы обеспечить доступ к нуклеиновым основаниям тех дНТФ, из которых ДНК-полимераза формирует дочернюю нить.
Это расплетание, используя энергию гидролиза АТФ, проводят ферменты геликазы.
Расплетание начинается со специфических последовательностей в ДНК, которые называются началом репликации (replication origin) или просто ориджином. Последовательности нуклеотидов в ориджинах разных организмов сильно варьируются, хотя они имеют и общее свойство - в ориджинах повышенная концентрация А-Т пар. В таких областях меньше водородных связей между нитями ДНК, и поэтому "легче" осуществлять "плавление" (расплетание) ДНК.
Как только геликаза расплетет дуплекс, особый фермент праймаза (специализированная РНК-полимераза) синтезирует короткий РНК-праймер, комплементарный расплетенному участку матричной цепи. Спаренный с ДНК праймер затем удлиняется ДНК-полимеразой, формируя дочернюю цепь.
Область ДНК, в которой собраны все эти ферменты для синтеза дочерних цепей, называется репликационная вилка или вилка роста.
В ходе репликации репликационная вилка и ассоциированные с нею белки удаляются от ориджина, что сопровождается ростом торсионных напряжений в ДНК. Эти напряжения снимаются ферментом mono-изомераза I. Для того чтобы процесс репликации протекал непрерывно и ДНК-полимераза перемещалась и копировала дуплекс ДНК, геликаза должна непрерывно расплетать дуплекс, а топоизомераза должна удалять образующиеся суперспирали.
Главная проблема при "обработке" ДНК репликационной вилкой вытекает из двух факторов:
1) две нити родительской ДНК антипараллельны,
2) ДНК-полимераза (так же, как и РНК-полимераза) катализирует рост дочерней цепи только в направлении 5'→3'.
Синтез дочерней цепи, которая называется лидирующая цепь, может происходить непрерывно, начиная с единственного праймера в 5'→3' направлении, то есть в том направлении, в котором перемещается репликационная вилка (рисунок 53).
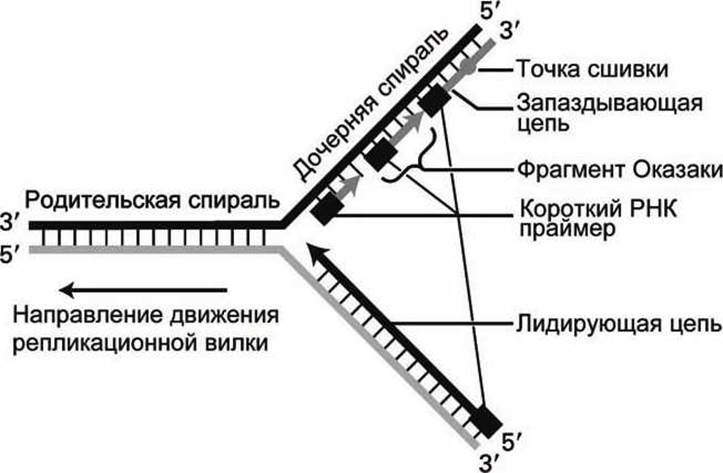
Рисунок 53 - Схема формирования лидирующей и запаздывающей цепей
Проблемы возникают при синтезе другой дочерней цепи, которая называется запаздывающей.
Поскольку рост запаздывающей цепи должен происходить в 5'→3' направлении, копирование матрицы должно осуществляться в направлении противоположном движению репликационной вилки.
Клетка решает эту проблему, синтезируя новые праймеры через каждые несколько сотен оснований на матричной цепи, по мере того, как дуплекс расплетается, и все более оснований освобождаются при расплетании.
Каждый из этих праймеров, спаренный с матричной цепью, удлиняется в 5'→3' направлении, образуя непрерывные сегменты, которые называются фрагменты Оказаки (в честь Рейджи Оказаки, который их открыл).
РНК-праймер каждого фрагмента Оказаки удаляется и замещается ДНК-цепью соседнего растущего фрагмента Оказаки. Фермент ДНК-лигаза сшивает соседние фрагменты.
Детальная информация о том, как белки участвуют в репликации ДНК эукариот, была получена благодаря исследованию репликации малых вирусных ДНК, в частности кольцевой ДНК вируса SV40, инфицирующего обезьян (рисунок 54).

Рисунок 54 - Схема репликационной вилки SV40 ДНК и ассоциированных белков: а - репликационная вилка; б - схема PCNA, охватывающего дуплекс ДНК; в - схема расположения RPA относительно матричной цепи ДНК
На рисунке 54 представлены те белки, которые координируют копирование SV40 ДНК в репликационной вилке. Этот многокомпонентный комплекс позволяет клетке реализовывать упорядоченную последовательность событий, которые обеспечивают важные функции клетки.
В молекулярной "машине", которая реплицирует SV40 ДНК, гексамер вирусного белка, который называется большой Т-антиген, выполняющий функцию геликазы, расплетает родительскую спираль в репликационной вилке.
Все остальные белки, которые участвуют в репликации, это собственные белки инфицированной клетки.
Праймеры для лидирующей и запаздывающей дочерних цепей ДНК синтезируются комплексом (праймаза-Pol а). В этом комплексе совместно работают праймаза, которая синтезирует короткий РНК-праймер, и ДНК-полимераза a (Pol а), которая наращивает РНК-праймер дезокси-нуклеотидами, образуя смешанный РНК-ДНК праймер.
Далее праймер наращивается в дочернюю нить ДНК ферментом ДНК-полимераза δ (Pol δ), которая точнее, с меньшим количеством ошибок, чем Pol а, копирует матричную цепь. В литературе ДНК-полимеразу δ ещё называют ДНК-полимераза III.
Pol δ формирует комплекс (PCNA-Rfc-Pol δ) с фактором репликации С, Rfc (replication factor С), и ядерным антигеном размножения клеток, PCNA (proliferating cell nuclear antigen), который заменяет комплекс (праймаза-Pol а) после синтеза праймазы.
Гомотримерный белок PCNA имеет центральный канал, через который проходит дочерний дуплекс ДНК (рисунок 54(6)). Тем самым PCNA предотвращает диссоциацию комплекса (PCNA-Rfc-Pol δ) с матричной цепи.
После того, как родительская ДНК разделилась на репликационной вилке на две матричные нити, с этими нитями связываются гетеротримерные белки RPA (replication protein А) (рисунок 54(b)). Белки RPA относятся к классу белков, связывающихся с однонитевой ДНК (single-strand binding proteins), которые ещё называют белками, которые дестабилизируют двойную спираль, (helix-destabilizing proteins).
Связывание с RPA поддерживает оптимальную конформацию матричной цепи для репликации ДНК-полимеразой. Эти RPA-белки отсоединяются от матричной цепи полимеразами Pol а и Pol δ по мере того, как они синтезируют комплементарные нити, спаренные с родительскими нитями.
Некоторые эукариотические белки, которые участвуют в репликации ДНК, не показаны на рисунке 54.
Фермент топоизомераза I связывается с родительской ДНК перед геликазой, чтобы устранять торсионные напряжения, которые возникают при расплетании спирали.
Специальные ферменты рибонуклеаза Н и белок FEN I удаляют рибонуклеотиды на 5'-конце фрагментов Оказаки. Эти рибонуклеотиды заменяются на дезоксирибонуклеотиды ДНК-полимеразой Pol δ, когда она достигает соседнего фрагмента Оказаки.
Завершенные фрагменты Оказаки сшиваются ферментом ДНК-лигаза, катализирующим образование стандартной 5'—3' фосфодиэфирной связи.
В ходе синтеза ДНК-полимераза приблизительно один раз на 10000 шагов ошибается и присоединяет неправильное основание к растущей ДНК-цепи. Геном человека содержит 3x109 пар оснований, и если бы не осуществлялся контроль и исправление ошибок синтеза, интенсивность возникновения мутаций была бы катастрофической. К счастью, ДНК- полимераза имеет встроенный механизм пруфридинга (proofreading, нахождение и исправление ошибок).
В ДНК полимеразе есть два активных центра.
Первый активный центр контролирует пространственное расположение и соответствие матричной цепи каждого следующего нуклеотида и катализирует синтез сахарофосфатной связи между новым нуклеотидом и уже синтезированным участком цепи ДНК.
После окончания "работы" первого активного центра второй активный центр оттягивает это последнее нуклеиновое основание, пытаясь оторвать его от синтезированной двойной цепи ДНК. Если связь нового основания недостаточно прочная - второй активный центр отрывает его и удаляет. Такой дополнительный этап устраняет нестандартное спаривание и в тысячи раз увеличивает точность матричного синтеза.