БИОХИМИЯ - Л. Страйер - 1984
ТОМ 3
Часть IV ИНФОРМАЦИЯ
ГЛАВА 26. ГЕНЕТИЧЕСКИЙ КОД Т ЗАВИСИМОСТЬ МЕЖДУ ГЕНАМИ И БЕЛКАМИ
26.4. Состав кодонов многих аминокислот был определен с помощью сополимеров в качестве матриц
Для дальнейшего изучения генетического кода были использованы в качестве матриц полирибонуклеотиды, состоящие из оснований двух типов. Например, случайный сополимер Uи G содержит восемь различных триплетов: UUU, ЦОС, UGU, GUU, UGС, GUG, GGU и GGG. Относительную частоту этих триплетов можно легко вычислить, исходя из молярного соотношения U и G в сополимере. Оно составляло 0,76:0,24 (табл. 26.1). Относительное включение различных аминокислот в присутствии этой матрицы приведено в табл. 26.2. В наибольшей степени включался, как и можно было предполагать, фенилаланин, так как триплет UUU встречался чаще всего. Затем шли валин, лейцин и цистеин. Уровень их включения составлял чуть больше трети от включения фенилаланина, что соответствует вычисленной частоте встречаемости триплетов, содержащих два U и одно G. Включение других аминокислот было очень низким. Отсюда был сделан вывод, что валин, лейцин и цистеин кодируются кодонами, содержащими 2U и 1G, а триптофан и глицин кодируются кодонами, которые содержат 1U и 2G.
Таблица 26.1. Ожидаемая встречаемость триплетов в случайном сополимере U (0,76) и G (0,24)

Таблица 26.2. Включение аминокислот в присутствии случайного сополимера U (0,76) и G (0,24)
Эксперименты того же типа проводили и с другими случайными сополимерами, а именно с UA, UC, АС и AG, а также с UGC, AGC, UAC и UAG. Таким образом в лабораториях Ниренберга и Очоа был определен состав кодонов, соответствующих каждой из 20 аминокислот.
26.5. Тринуклеотиды способствуют связыванию определенных молекул тРНК с рибосомами
При использовании смешанных сополимеров в качестве матриц удалось установить состав кодонов, соответствующих определенным аминокислотам, но не их последовательность (кроме UUU, ААА и ССС). Как уже говорилось, валин кодируется триплетом из 2U и 1G. Какой же это триплет: UUG, UGU или GUU? Ответ на этот вопрос был получен с помощью двух совершенно различных экспериментальных подходов: во-первых, с использованием синтетических полирибонуклеотидов с упорядоченной последовательностью и, во- вторых, с помощью зависимого от кодона специфического связывания молекул тРНК с рибосомами.
В 1964 г. Ниренберг установил, что тринуклеотиды способствуют связыванию определенных молекул тРНК с рибосомами в отсутствие синтеза белка. Например, добавление pUpUpU вызывает связывание фенилаланиновой тРНК, а рАрАрА значительно увеличивает связывание лизиновой тРНК, как и рСрСрС - связывание проли- новой тРНК. Динуклеотиды не стимулируют связывания тРНК с рибосомами. Эти работы показали, что тринуклеотид (как и триплет в мРНК) специфически связывается с определенной молекулой тРНК, для которой он является кодовым словом. Был разработан простой и быстрый тест на связывание: связанные с рибосомами молекулы тРНК задерживаются на нитроцеллюлозном фильтре, а несвязанные молекулы тРНК проходят через фильтр. Для того чтобы определить, какие именно молекулы тРНК садятся на фильтр, использовали молекулы тРНК, несущие определенную аминокислоту, меченную С.
С помощью методов органической химии и биохимии были синтезированы все 64 тринулеотида. Для каждого тринуклео- тида было проверено связывание молекул тРНК, соответствующих всем 20 аминокислотам. Например, было показано, что pUpUpG стимулирует связывание только лейциновой тРНК, pUpGpU - только цистеиновой тРНК, a pGpUpU - связывание только валиновой тРНК. Отсюда был сделан вывод, что кодоны UUG, UGU и GUU соответствуют лейцину, цистеину и валину соответственно. С несколькими кодонами не было получено избирательного связывания какой-либо тРНК, тогда как с несколькими другими связывалось более одной тРНК. Для большинства кодонов были получены вполне четкие результаты.
В общем этот простой и элегантный подход позволил расшифровать около 50 кодонов.
26.6. Еще один инструмент расшифровки кода - сополимеры с определенной последовательностью
Примерно в то же время Гобинду Коране (Gobind Khorana) удалось синтезировать полирибонуклеотиды с определенной повторяющейся последовательностью. Сочетая методы органической химии и ферментативные методы, он синтезировал ряд сополимеров с повторяющейся последовательностью из двух, трех и четырех оснований. Рассмотрим, к примеру, стратегию синтеза poly(GUA). Этот упорядоченный сополимер имеет последовательность
GUAGUAGUAGUAGUAGUAGUAGUA...
Прежде всего Корана синтезировал с помощью методов органической химии два комплементарных дезоксирибонуклеотида по девять нуклеотидов длиной: d(TAC)3 и d(GTA)3. Затем эти два олигонуклеотида были использованы в качестве матрицы для синтеза длинных цепей ДНК из четырех дезоксинуклеозидтрифосфатов под действием ДНК-полимеразы I. Ни один олигонуклеотид в отдельности не был эффективной матрицей. Если же присутствовали оба олигонуклеотида, то d(TAC)3 служил матрицей для синтеза poly(dGTA), a d(GTA)3 - для синтеза poly(dTAC). Эти длинные комплементарные ДНК образовывали двухспиральные молекулы. Следующим шагом было получение длинных полирибонуклеотидных цепей с последовательностью, соответствующей poly (dTAC) и poly(dGTA). Для этого дуплекс poly(dTAC): poly(dGTA) использовали в качестве матрицы для РНК-полимеразы. Цепь ДНК для транскрипции можно выбрать, добавляя три подходящих рибону- клеозидтрифосфата. Если в инкубационную смесь добавить GTP, UTP и АТР, го на матричной цепи poly(dTAC) синтезируется полирибонуклеотидный продукт poly(GUA). Другая цепь не транскрибируется, так как не хватает одного из необходимых субстратов - СТР. Если же добавить СТР, UTP и АТР, то на другой матричной цепи синтезируется poly(UAC). Итак, органический синтез и вслед за ним матричный синтез с помощью ДНК-полимеразы и РНК-полимеразы позволили синтезировать два длинных полирибонуклеоти- да со строго определенной повторяющейся последовательностью оснований (рис. 26.4).
Рис. 26.4. Цепь этой двухспиральной матричной ДНК, которая должна транскрибироваться, определяется набором рибонуклео- зидтрифосфатов в инкубационной смеси

Эти регулярные сополимеры были использованы в качестве матриц в бесклеточной системе синтеза белка. Рассмотрим некоторые результаты такого эксперимента. Сополимер, состоящий из чередующейся последовательности двух оснований - А и Б:
![]()
содержит кодоны двух видов - АБА и БАБ. Поэтому полипептидный продукт должен представлять собой чередующуюся последовательность из двух аминокислот (сокращенно обозначенных ак1 и ак2):
![]()
Стоит ли на N-конце полипептидного продукта ak1 или ак2, зависит от того, начинается ли рамка считывания с А или с Б. Если в качестве матрицы использовали poly(UG), то синтезировался полипептид из чередующихся валина (Val) и цистеина (Сvs):
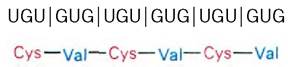
Этот результат однозначно доказывал три- плетность кода и показывал, что один из триплетов - UGU или GUG - кодирует цистеин, а другой - валин. В сочетании с данными по связыванию тРНК было очевидно, что UGU кодирует цистеин, a GUG - валин. В присутствии некоторых чередующихся сополимеров из двух оснований происходил синтез следующих полипептидов:

Рассмотрим теперь матрицу, состоящую из повторяющейся последовательности трех оснований, поли(АБВ). Если рамка считывания начинается с А, образующийся полипептид должен содержать аминокислоту только одного вида, кодируемую триплетом АБВ:

Если рамка считывания начинается с Б, синтезирующийся полипептид должен содержать другую аминокислоту, кодируемую триплетом БВА:
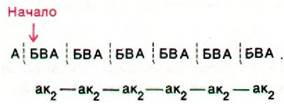
Если же рамка считывания начинается с В, должен образовываться полипептид третьего типа, содержащий аминокислоту, кодируемую триплетом ВАБ:
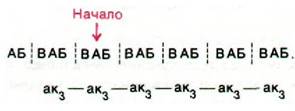
Таким образом, предполагаемые продукты - три различных гомополипептида. Действительно, именно такой результат получался в случае большинства матриц, состоящий из повторяющейся последовательности трех нуклеотидов. Например, на poly(UUC) синтезировались полифенилаланин, полисерин и полилейцин. Этот результат в сочетании с результатами других экспериментов показывал, что UUC кодирует фенилаланин, UCU-серин и CUU-лейцин. Полипептиды, которые синтезировались на других матрицах этого типа, приведены в табл. 26.3. Обратите внимание, что в присутствии poly(GUA) и poly(GAU) происходил синтез двух, а не трех гомополипептидов. Причина этого явления станет ясна чуть ниже.
Таблица 26.3. Гомополнпептиды, синтезирующиеся на матрицах, которые состоят нз повторяющихся последовательностей тринуклеотидов
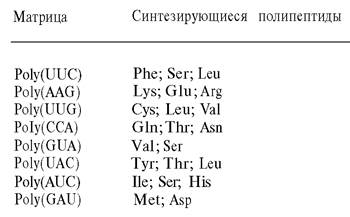
Корана синтезировал также ряд сополимеров, состоящих из повторяющегося тетрануклеотида, например, poly(UAUC). На этой матрице шел синтез полипептида с повторяющейся последовательностью Tyr-Leu-Ser-Ile независимо от рамки
считывания:
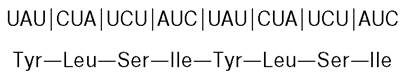
Отсюда можно было сделать вывод о смысле четырех кодонов.
Совершенно иной результат был получен при использовании в качестве матрицы poly(GUAA). Единственными продуктами были ди- и трипептиды. Почему не было более длинных цепей? Объясняется это тем, что один из триплетов, встречающихся в этом сополимере, а именно UAA, кодирует не аминокислоту, а терминацию синтеза белка:
![]()
На матрице poly(AUAG) также получались только ди- и трипептиды, так как UAG- второй сигнал терминации цепи:
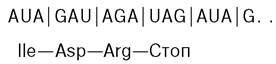
Посмотрим теперь снова на табл. 26.3. На матрице poly(GUA) синтезировались два, а не три гомополипептида по той причине, что третья рамка считывания соответствует последовательности

т. е. представляет собой повторяющуюся последовательность сигнала терминации. Как же обстоит дело с poly(GAU)? На этой матрице синтезировались только два гомополипептида, потому что третья рамка считывания соответствует еще одному сигналу терминации - UGA:
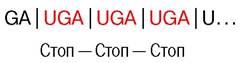
Синтез полинуклеотидов с определенной последовательностью в лаборатории Кораны был выдающимся достижением. Использование этих полимеров в качестве матриц для синтеза белка в сочетании с работами Ниренберга по связыванию тРНК с рибосомами в присутствии тринуклеотидов привели к полной расшифровке генетического кода к 1966 г. Еще за шесть лет до этого такое событие казалось несбыточной мечтой. Благодаря разработке совершенных методов синтеза в лаборатории Кораны стало возможным и еще одно достижение: полный синтез молекулы ДНК, соответствующей последовательности молекулы транспортной РНК.