Основы биоинформатики - Огурцов А.Н. 2013
Информационные принципы в биотехнологии
Секвенирование биологических последовательностей и экспрессия генов
Ярлыки экспрессируемых последовательностей
Ярлык экспрессируемой последовательности EST (Expressed Sequence Тags) - это секвенированный отрезок последовательности клона, случайно отобранного из библиотеки кДНК, используемый для опознавания генов, экспрессируемых в определённой ткани.
Мы далеко не всегда располагаем расшифровками полных последовательностей ДНК; в основном накопленные к настоящему времени данные о ДНК состоят из отдельных отрезков последовательностей, большая часть которых представлена ярлыками экспрессируемых последовательностей (EST).
В анализе EST-последовательностей необходимо учитывать следующие моменты:
1) алфавит EST-последовательностей состоит из пяти знаков (а, с, g, t, u)
2) в последовательности могут присутствовать фантомные всуды (сокр. вставка/удаление - insdel, от англ. insertion/deletion - инсерция/делеция), приводящие к сдвигам рамки трансляции;
3) весьма вероятно, что EST-последовательность окажется подпоследовательностью какой-либо последовательности из баз данных;
4) EST-последовательность может вовсе не представлять отрезок кодирующей последовательности (coding sequence) какого-либо гена.
Принцип секвенирования EST-последовательностей показан на рисунке 69.
Из клеток интересующей ткани или клеточной линии создают библиотеку кДНК. Для этого из ткани или культуры клеток выделяют мРНК. Затем мРНК обратно транскрибируют в кДНК — обычно с помощью праймера олиго-(дТ), так что один конец встройки кДНК получается транскрибированным с поли-А хвоста на конце мРНК. Другой конец кДНК обычно соответствует некоторому участку кодирующей последовательности или, если кодирующая последовательность коротка, - участку 5'-EST. Наконец, полученную кДНК клонируют с помощью вектора.

Рисунок 69 - Схема конструирования EST - ярлыков экспрессируемой последовательности: 1 - выделение мРНК и обратная транскрипция в кДНК; 2 - встраивание кДНК в вектор для размножения и создания библиотеки кДНК;
3 - отбор отдельных клонов; 4 - секвенирование 5'- и 3'-концов встроенной кДНК; 5 - помещение EST в базу данных dbEST
Отдельные клоны выбирают из библиотеки и синтезируют по одной последовательности с каждого конца встройки кДНК. Такой метод называют RACE-методом (Rapid Amplification of С-DNA Ends - быстрая амплификация концов кДНК).
Таким образом, каждый клон обычно представлен 5'-EST и З'-EST. Поскольку EST-последовательности коротки, то они обычно представляют только фрагменты генов, а не полные кодирующие последовательности. Типичный ярлык EST имеет длину от 200 до 500 нуклеотидов.
Как правило, процесс синтеза ярлыков EST в высокой степени автоматизирован и обычно предполагает использование флуоресцентной лазерной системы для считывания гелевых плёнок. Дальнейший анализ расшифрованных последовательностей загружаются производится компьютерными методами.
Для выяснения, представляет ли такая EST-последовательность новый ген, проводят поиск в базе данных ДНК. Если результат выравнивания показывает существенное подобие с некоторой последовательностью в базе данных, то нормальная процедура классификации совпадений определит, был ли найден действительно новый ген.
Если, однако, результат поиска не показывает значительного подобия, то мы не имеем достаточных оснований предполагать, что был обнаружен новый ген. Может оказаться и так, что данная EST-последовательность представляет некодирующую последовательность какого-либо известного гена, которую просто не успели поместить в базу данных.
Во многих мРНК (особенно у человека) на 5'- и 3'-концах кодирующей последовательности расположены длинные нетранслируемые области UTR (рисунок 43). Весьма вероятно, что данная EST-последовательность была целиком транскрибирована с одной из этих некодирующих UTR-областей. Если нам повезет, то в базе данных уже будет находиться некоторые фрагменты нетранслируемой (некодирующей) последовательности. Если это так, то при поиске будет найдено прямое совпадение, поскольку нетранслируемые области UTR сильно консервативны и весьма специфичны к кодирующим генам.
В случае неблагоприятного исхода не будет найдено никакого совпадения, что указывает на одну из следующих двух возможностей:
1) данная EST-последовательность представляет некоторую кодирующую последовательность, для которой нет ни одной подобной последовательности в базе данных;
2) эта EST-последовательность представляет некодирующую последовательность, которая ещё не помещена в базу данных.
В интерпретации анализа EST-последовательностей важно чётко различать эти две ситуации
На рисунке 70 представлена схема соответствия ДНК, кДНК и EST.
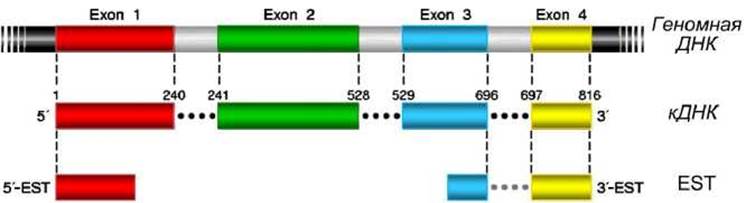
Рисунок 70 - Выравнивание полностью секвенированных последовательностей кДНК и ярлыков EST с геномной ДНК
Точки между сегментами кДНК или EST-последовательностями обозначают области в геномной ДНК, которые не выравниваются с последовательностями кДНК или EST - это области нитронов.
Числа над линией кДНК обозначают координаты (в нуклеотидах) последовательности кДНК, где нуклеотид №1 - ближайший к 5'-концу кДНК, а нуклеотид №816 - ближайший к 3'-концу кДНК.
Каждый ярлык EST представляет только короткую последовательность, считанную с 5'- или с 3'-конца соответствующей кДНК.
Таким образом, ярлыки EST устанавливают границы единиц транскрипции, но не дают никакой информации о внутренней структуре транскриптов, если только последовательности этих EST не пересекают нитроны (как в случае З'-EST, изображённой на рисунке 70).